class: center, middle, inverse, title-slide # Programmieren Mit R ### Christoph Rust ### 06.11.2019 --- # Übersicht über den Kurs 0. Vorab 1. Einführung 2. Objekte in R 3. Graphiken 4. Datenanalyse (dplyr) 5. Programmieren: Flow Control 6. Regressionen 7. Monte Carlo Simulationen 8. Numerische Optimierung 9. Bootstrap 10. Effizienz --- # 0. Vorab ### Infrastruktur - Privater Laptop mit R und RStudio - CIP-Pool-Rechner - Ordnerstruktur: + code/ + RData/ ### Literatur/Links - Ligges, U. (2008), [Programmieren mit R](https://www.springer.com/de/book/9783540799979), Springer. - Kleiber, C. & Zeileis, A. (2008), [Applied Econometrics with R](https://www.springer.com/de/book/9780387773162), Springer. - Braun, J. & Murdoch, D.(2007), [A first course in statistical programming with R](http://einspem.upm.edu.my/wopr2017/2016.pdf) - Hadley Wickham, [Advanced R](https://adv-r.hadley.nz/) - Kurshomepage: [Programmieren mit R](http://www.uni-regensburg.de/wirtschaftswissenschaften/vwl-tschernig/lehre/master/programmieren-mit-r/index.html) - [R Website](https://www.r-project.org/) --- # 0. Vorab ### Lernziele: "Programmieren in der Ökonometrie" - Grundlagenverständnis für R - "Hilfe zur Selbsthilfe" - Umgang mit und Auswertung von Daten (Deskriptive Statistiken, Visualisierung) - Einfache Modellschätzungen - Programmiergrundkenntnisse - Monte-Carlo-Simulationen und Bootstrap (Basis für MCMC) - Optimierung ### Klausur - Freitag, 10.01.2020, 10:00 Uhr - Dauer: 60 Minuten - Raum: CIP-Pool SG1 - R-Skript mit zu lösenden Problemen - Anmeldung per E-Mail (siehe [Kurshomepage](http://www.uni-regensburg.de/wirtschaftswissenschaften/vwl-tschernig/lehre/master/programmieren-mit-r/index.html)) --- # 0. Vorab ### Installation 1. Download von einem "CRAN" Spiegelserver z.B. [https://ftp.fau.de/cran/](https://ftp.fau.de/cran/) 2. Auf "Download R for {Windows/OS X/Linux)" klicken, danach auf "base". 3. Installation abhängig vom System. Bei vielen Linux-Distributionen häufig in Paketquellen verfügbar. 4. R starten. --- # 1. Einführung ### Allgemeines **R** ist... - eine freie Implementierung der Programmiersprache **S** mit den Zielen: + Interaktives Rechnen mit Daten + Den Benutzer einfach zum Programmierer werden zu lassen + Erstellen von Graphiken zur Datenanalyse + Einfache Wiederverwendbarkeit von vorhandenen Funktionen - eine (vom R-Interpreter) bei Laufzeit *interpretierte* Sprache - eine *funktionale* Sprache (Funktionen sind first-level Objekte) - eine *objektorientierte* Sprache (Klassen und Methoden) - eine *vektorisierte* Sprache (Objekte werden intern als Vektoren repräsentiert) --- # 1. Einführung ### Vorteile von R - eine freie (open-source, GPL2/3) Software, jeder Code kann damit eingesehen und überprüft werden - sehr nah an der (statistischen) Forschung - leicht durch Pakete erweiterbar - auf fast allen Plattformen lauffähig ### Nachteile von R - keine grafische Benutzeroberfläche (aber R-Studio) - keine interaktiven Grafiken (aber Shiny) - interpretierte Sprache, daher im vergleich zu kompilierten Sprachen manchmal langsam. Kompilierter Code (C/C++, Fortran, Rust) lässt sich aber einbinden, um dies zu umgehen. --- # 1. Einführung ### Erste Schritte - Eingabe von *expressions* in die R-Konsole (der Interpreter), Ausführen mit *Return* - mit Pfeiltasten <em>(↑, ↓)</em> lassen sich wieder zuletzt eingegebene *expressions* holen ```r > sin(0) > 2 - 1 > 0/0 # -> NaN (nicht definiert) > Inf-Inf # -> NaN (nicht definiert) > 2 + 3*4 # Punkt vor Strich > 2 + + 1 # Ausführung erst wenn expression vollständig ``` ``` ## [1] 0 ## [1] 1 ## [1] NaN ## [1] NaN ## [1] 14 ## [1] 3 ``` --- # 1. Einführung Grafiken: ```r set.seed(123) x <- runif(100) y <- x + rnorm(100, sd = 0.1) plot(x,y) ``` <img src="pmr-intro_files/figure-html/list2-1.png" style="display: block; margin: auto;" /> --- # 1. Einführung Mit der Kommandozeile sind zwar eine Menge einfacher Berechnungen möglich, sobald es aber komplizierter wird, sollten *Skripte* genutzt werden. Ein Skript ist eine Textdatei (in der Regel endet der Dateiname mit '.R'), welche R-Code enthält. Z.B: ```r ## Arbeitsverzeichnis wechseln setwd("C:/Users/Max/R-Code") ## Daten laden myData <- read.table("all_important_data.csv", sep = ";", header = TRUE) ## Summary summary(myData) ``` --- # 1. Einführung ### Editoren Textdateien werden mit Texteditoren bearbeitet, es gibt einige Editoren welche die Arbeit mir R vereinfachen: - R-Studio (in diesem Kurs verwendet) + Kommt einer GUI am nächsten + Plot, Übersicht über Objekte und Pakete in einem Fenster + Extra-Klick-Funktionen wie Daten laden + gerade für Anfänger sehr zu empfehlen - Notepad++ in Verbindung mit npptor (Notepad++ to R) + Flexibler Editor für alle möglichen Programmiersprachen, txt-Files etc. (ein Editor für mehrere Programmiersprachen?) + Extrem funktional als Editor: Add-Ons, Makros, Hotkeys etc. + Einrückungen, Hervorhebungen, Aus- und Einkommentierungen komfortabel --- # 1. Einführung - (X)Emacs mit ESS + Extrem vielseitiger mächtiger Editor der allerdings etwas Einarbeitung erfordert + beliebig konfigurierbar + läuft auf allen Plattformen + Editor eignet sich für alle möglichen Anwendungsfälle (LaTeX, email, git,...) --- # 1. Einführung ### RStudio - Struktur: 4 Fenster + links oben: Code-Editor, data.frame-View (**Strg + 1**) + links unten: R-Konsole (**Strg + 2**) + rechts oben: Anzeige der Objekte in der globalen Umgebung (**Strg + 8**), History (**Strg + 4**) + rechts unten: Hilfe (**Strg + 3**), Plots (**Strg + 6**), Pakete (**Strg + 7**),... - Einzelne Fenster können inzwischen auch vom Hauptfenster losgelöst werden - Um Code aus dem Editor auszuführen: + Curser in die jeweilige Zeile setzen + **Strg + Enter** drücken `\(\rightarrow\)` Curser springt in die nächste Zeile --- # 1. Einführung ### Hilfe zu einem Thema Um Hilfe zu einer *bekannten* Funktion zu erhalten kann man entweder im RStudio-Hilfe-Tab nach der Funktion suchen oder in der R-Konsole ```r ?getwd ``` eingeben:<br> <img src="figures/getwd-help.png" style="width: 60%" align="center" /> --- # 1. Einführung ### Hilfe zu einem Thema Sucht man beispielsweise eine *unbekannte* Funktion (z.B. eine Funktion, die den t-Test durchführt) dann am besten Google nutzen, z.B.: <img src="figures/google-search.png" style="align:center;width: 60%" /> --- # 1. Einführung ### Hilfe zu einem Thema - Jedes Paket hat eine eigene Dokumentation (enhält die Hilfen), häufig Vignetten (etwas ausführlichere Erklärungen) - stackoverflow - Mailinglisten R-help --- # 1. Einführung ### R als Taschenrechner ```r 1 + 2 # -> 3 1 + (2 * 4) # -> 9 a <- 3 b <- 3 * a # -> 9 sqrt(b) # -> 3 ``` <img src="figures/basic-operations.png" style="align:center;width: 60%" /> --- #1. Einführung ### Funktionen Immer wenn etwas *passiert*, werden Funktionen aufgerufen. Alle Operatoren, control-flow,..., sind in R Funktionen. - Ein Funktionsaufruf geschieht dadurch, dass der Funktionsname angegeben wird und in runden Klammern ein oder mehrere durch Kommata getrennte Argumente folgen: `funktionsname(Argument1 = Wert1, Argument2 = Wert2,...)` - Die Argumentnamen müssen nicht zwingendermaßen angegeben werden: `funktionsname(Wert1,Wert2,...)` - Achtung: Reihenfolge relevant! - Es gibt auch voreingestellte Argumente, diese müssen auch nicht angegeben werden. Dazu später mehr. --- #1. Einführung ### Zuweisung Die Zeichenkombination `<-` ist der Zuweisungsoperator und speichert den Wert des Ausdruckes rechts von ihm in die Variable links ```r a <- (3 * 4) (3 * 4) -> a # auch möglich aber nicht zu empfehlen a = (3 * 4) # auch häufig verwendet aber nicht äquivalent zu '<-' a<-(3*4) # schlecht lesbar ``` - Der Lesbarkeit halber sollten um den Zuweisungsoperator immer Leerzeichen stehen. - Variablennamen müssen mit einem Buchstaben beginnen, dürfen aber auch Zahlen, Punkt und Unterstrich enthalten - Es empfiehlt sich eine einheitliche Benennung der Objekte, später dazu mehr. --- # 1. Einführung ### Logische Operatoren <img src="figures/logical-operators-values.png" style="align:center;width: 60%" /> Beispiele ```r TRUE & FALSE # FALSE TRUE & TRUE # TRUE TRUE | FALSE # TRUE !TRUE | FALSE # FALSE FALSE && TRUE # zweites TRUE wird nicht ausgewertet TRUE && TRUE # zweites TRUE wird ausgewertet TRUE || FALSE # zweites FALSE wird nicht ausgewertet ``` --- # 1. Einführung Weitere Beispiele ```r c(TRUE, FALSE) & c(TRUE, TRUE) # [1] TRUE FALSE -> Vektorwertig c(TRUE, FALSE) && c(TRUE, TRUE) # [1] TRUE -> nicht Vektorwertig ``` Quantoren: ```r a <- c(TRUE, FALSE, TRUE) b <- c(TRUE, TRUE, TRUE) any(a) # [1] TRUE all(a) # [1] FALSE all(b) # [1] TRUE ``` --- # 1. Einführung Fehlende Werte: Testen auf `NA` nur mit `is.na()` möglich. `x == NA` liefert immer `NA`! Beispiele: ```r a <- c(TRUE, NA, TRUE) a == NA # [1] NA NA NA is.na(a) # [1] FALSE TRUE FALSE ``` --- # 1. Einführung ### Weitere nützliche Funktionen: - `ls()` zeigt die vorhandenen Objekte in der globalen Environment - `str()` zeigt die Struktur eines Objektes an - `rm()` löscht ein Objekt aus dem globalen Workspace - `getwd()` zeigt das aktuelle Arbeitsverzeichnis an - `setwd()` wechselt das Arbeitsverzeichnis + Unter Windows ist der Pfadtrenner entweder `/` oder `\\` + Unter Linux/Mac immer `/` - `save()` speichert Objekte als `.RData`-Datei ab. - `load()` lädt Objekte in die globale Environment - `list.files()` zeigt Dateien im angegebenen Verzeichnis an - `source()` führt ein R-Skript aus --- # 1. Einführung ## Erweiterbarkeit durch R-Pakete Es gibt auf [CRAN](https://cran.r-project.org/) eine Vielzahl verschiedenster Pakete für alle möglichen Anwendungen. Damit lässt sich das (relative kleine) Grundsystem beliebig erweitern. R wird mit einigen Standard-Paketen ausgeliefert viele müssen aber nachträglich erst installiert werden. ```r install.packages("AER") # installiere ein bisher nicht vorhandenes Paket library(AER) # Binde Paket ein (macht Objekte aus dem Paket im # globalen Namensraum sichtbar) data(CASchools) # z.B. Datensatz "CASChools" ?ivreg # Hilfe zur Funktion ivreg ``` - `search()` zeigt den Suchpfad an, also auch welche Pakete bereits geladen sind: ```r search() ``` ``` ## [1] ".GlobalEnv" "package:stats" "package:graphics" ## [4] "package:grDevices" "package:utils" "package:datasets" ## [7] "package:methods" "Autoloads" "package:base" ``` --- # 1. Einführung ## Aufgabe 1.1 Erzeugen Sie eine Datei `test.R` in ihrem codes-Ordner. Diese enthält ein Skript, das das Objekt `x`, belegt mit der Zahl `5`, und das Objekt `y`, belegt mit der Zahl `6`, erzeugt. Bevor Sie diese `test.R`-Datei mit der `source`-Funktion aufrufen, löschen Sie Ihren gesamten Workspace. Sehen Sie sich danach den Workspace an, berechnen Sie das Produkt der beiden Zahlen, löschen Sie danach das Objekt `x`, speichern Sie den restlichen Workspace im ".RData"-Format im Ordner "RData". Hinweis: Allgemein ist es schlau, das Arbeitsverzeichnis nicht hin- und herzusetzen, sondern nur den Source-Befehl auf den expliziten Code-Ordner anzuwenden. ## Aufgabe 1.2 Testen Sie R als Taschenrechner: 1. Berechnen Sie den Wert der Sinusfunktion an der Stelle 0 2. Definieren Sie x als die Zahl 2 und berechnen sie die doppelte dritte Potenz von x. --- # 1. Einführung ## Aufgabe 1.3 Suchen Sie nach einem R-Paket, welches Funktionen bereitstellt um lineare Hypothesen im multiplen Regressionsmodell zu überprüfen. Installieren Sie das Paket und zeigen Sie die Hilfe zu einer Funktion an. --- # 2. Objekte ## Objekte in R - Alles was in R existiert (Objekte) ist entweder eine Daten(-struktur) oder eine Funktion - Objekte haben jeweils eine Klasse (Objektorientierung) ### Erstes Wichtiges Objekt: Funktionen Eine Funktion ist ein Programmkonstrukt, das eine Prozedur auf bereitgestellten Objekten ausführt und ein Ergebnis zurückgibt. ```r log(2.3) sin(2) class(log) class(2.3) ``` ``` ## [1] 0.8329091 ## [1] 0.9092974 ## [1] "function" ## [1] "numeric" ``` --- # 2. Objekte ## Funktionen Es gibt Funktionen mit und ohne Seiteneffekt: - Funktionen ohne Seiteneffekt nehmen Objekte entgegen und führen damit eine Operation aus dessen Ergebnis zurückgegeben wird. Beispiel: `log()` - Funktionen mit Seiteneffekt verändern darüber hinaus Objekte im globalen Workspace. Beispiel: `setwd()`, `'<-'()` (Zuweisung ist auch eine Funktion!) `\(\Rightarrow\)` Beim Entwickeln von Funktionen sollten Seiteneffekte möglichst vermieden werden (es sei denn, sie sind ausdrücklich erwünscht) --- # 2. Objekte ## Funktion definieren Eine Funktion wird folgendermaßen definiert: ```r ## einfache Funktion produkt1 <- function(x1, x2) x1 * x2 ## mit vorbelegten Argumenten produkt2 <- function(x1 = 1, x2 = 2) x1 * x2 ## häufig geschweifte Klammer um Funktionskörper produkt3 <- function(x1 = 1, x2 = 2){ x1 * x2 } ``` - Im obigen Beispiel sind geschweifte Klammern nicht nötig, sobald die Funktion aber mehrere Operationen ausführt sollten Sie verwendet werden --- # 2. Objekte ### Funktionen aufrufen ```r f1 # Funktionsdefinition anzeigen lassen # (print-Methode auf eine Funktion) f1() # Funktion aufrufen, allerdings verlangt # diese Funktion Argumente f1(x1=2, x2=5) f1(x1=2, x2=5, y=5) # Funktion akzeptiert exakt 2 Argumente, # 3 erzeugen darum einen Fehler ``` #### Was passiert beim Aufruf der Funktion? `f1(x1 = 1,x2 = 2)` Während der Laufzeit der Funktion wird eine neue Umgebung erzeugt, in dieser sind zwei Variablen vorhanden, x1 hier mit dem Wert 1, x2 hier mit dem Wert 2. Diese werden benutzt um das Produkt zu berechnen, dieses wird schließlich zurückgegeben. --- # 2. Objekte Nicht alle Funktionen verlangen Argumente, z.B. `getwd()` ```r f2 <- function() x1 * x2 x1 <- 2 x2 <- 3 f2 # Funktionsdefinition anzeigen lassen f2() # Funktion aufrufen f2(x1=2) # Erzeug einen Fehler, f2 akzeptiert kein Argument f2(2) ``` f2 hat keine Argumente. Das heißt aber auch, dass bei Laufzeit in der erzeugten Umgebung auch nicht die Variablen x1 und x2 sichtbar sind. In R ist die Regel, dass dann in der nächsthöheren Umgebung nach den Variablen x1 und x2 gesucht wird (also bspw. in der Umgebung aus der die Funktion aufgerufen wurde. Existiert x1 und x2 in keiner Ebene, führt das zu einem Fehler: ```r rm(x1,x2) f2() # Fehler ``` --- # 2. Objekte Beispiel etwas kompliziertere Funktion ```r f4 <- function(x1,x2) { z <- x1 + x2 abc <- x1/z return(abc) # return gibt Funktionswert aus und beendet Funktion abc <- 123 # von Funktion nicht mehr ausgeführter Code } f4(1,2) # -> 0.3333333 ``` --- # 2. Objekte Ein spezielles Argument ist das "Dreipunkte"-Argument (`...`). Damit lassen sich flexibel Argumente "durchreichen": ```r f5 <- function(e, ...) { log(e, ...)^2 } ``` alle Argumente, mit denen `f5` nichts anfangen kann, werden an `log()` weitergereicht: ```r f5(5) log(5, base=10)^2 # log() hat auch ein Argument "arg" f5(5, base = 10) # wird hier durchgereicht f5(5, base = 10, arg3 = "a") # mit arg3 kann aber log() auch nichts anfangen... ``` --- # 2. Objekte Es gibt ferner Funktionen, die Methoden aufrufen, insbesondere `print()`, `summary()`, `plot()`. Diese führen abhängig von der Klasse des übergebenen Objekts unterschiedliche Operationen aus. ```r plot(cos, -1, 1) # 'plot' Angewandt auf Funktion 'cos' x <- 1:5 y <- 6:10 plot(x, y) # 'plot' Angewandt auf zwei Vektoren 'x' und 'y' ``` Es gibt zwei (neuerdings auch drei) Klassensysteme in R (S3, S4 und Reference-Klassen, letzere am nächsten an OOP). --- # 2. Objekte ### Zusammenfassung Funktionen Alle Objekte die nicht Daten sind sind eine Funktion. Was man nun wissen sollte: Funktionsname, Funktionskörper, Funktionsdefinition Für den Quellcode einer Funktion (R ist KEINE Blackbox!) ist Uwe Ligges [Artikel in R news (p. 43)](https://cran.r-project.org/doc/Rnews/Rnews_2006-4.pdf) eine gute Referenz. "When looking at R source code, sometimes calls to one of the following functions show up: `.C()`, `.Call()`, `.Fortran()`, `.External()`, or `.Internal()` and `.Primitive()`. These functions are calling entry points in compiled code such as shared objects, static libraries or dynamic link libraries. Therefore, it is necessary to look into the sources of the compiled code, if complete understanding of the code is required. ... The first step is to look up the entry point in file `$R HOME/src/main/names.c`, if the calling R function is either `.Primitive()` or `.Internal()`." --- # 2. Objekte ### Aufgabe 2.1 1. Erzeugen Sie eine Funktion mit Namen "getSquaredSum", die bei Eingabe zweier Zahlen die quadrierte Summe der Zahlen berechnet und zurückgibt. Testen Sie diese Funktion mit zwei Möglichkeiten. Belegen Sie nun das zweite Funktionsargument mit 0 vor. 2. Erzeugen Sie eine Funktion in deren Funktionskörper eine weitere Funktion definiert und dann aufgerufen wird. Definieren Sie nun die "innere Funktion" außerhalb der äußeren. Was gefällt Ihnen besser? Überprüfen Sie, ob alles funktioniert und gleiche Werte liefert! --- # 2. Objekte ### Atomare Datentypen Aus atomaren Datentypen bestehen alle komplexeren Datentypen (Vektoren, Matrizen, Listen,...) - `NULL`: die leere Menge - `logical`: logische Werte - `integer`: ganze Zahlen - `numeric`: reelle Zahlen - `complex`: komplexe Zahlen - `character`: Buchstaben und Zeichenfolgen - für weitere siehe `?typeof` ```r typeof(2.3) # numeric typeof(TRUE) # logical typeof("abc") # character typeof(log) # special ``` --- # 2. Objekte ### Atomare Datentypen Für die atomaren Datentyp existiert jeweils eine Funktion, um zu überprüfen ob ein Objekt von diesem Datentyp ist sowie eine Funktion, die (falls sinnvoll möglich) in den entsprechenden Datentyp umwandelt: ```r is.numeric(1) # TRUE as.numeric("2.3") # 2.3 as.character(2.3) # "2.3" is.null(NULL) # TRUE ``` --- # 2. Objekte ## Vektoren Vektoren sind die grundlegende Struktur in R und besteht aus mehreren Elementen eines atomaren Datentyps. Mit der Funktion `c()` lassen sich Vektoren erzeugen: ```r x <- c(1, 2.3, 5, 1) x <- c(2, 2, 2, x) # neuer Vektor aus altem Vektor y <- c("Test", "Hallo") y <- c(y, x) # alles wird zu 'character', dem "niedrigsten" Datentyp x <- c(x, NA) # aber: NA verändert den Typ nicht! ``` --- # 2. Objekte Weitere Möglichkeiten um Vektoren zu erzeugen: - ganzzahlige Folgen: `1:5`, alternativ: `seq(1,5)` - beliebige Folgen: `seq(start, end, by)` - Wiederholungen: `rep()` ```r 2:4 # -> 2,3,4 seq(2,8,2) # -> 2,4,6,8 rep(2, 4) # -> 2,2,2,2 x <- 1:3 rep(x, 2) # -> 1,2,3,1,2,3 rep(x, each = 2) # -> 1,1,2,2,3,3 ``` Die einzelnen Elemente von Vektoren können auch benannt werden: ```r x <- c(eins = 2.4, zwei = 3, drei = 4, letztes = 2) ``` --- # 2. Objekte ### Rechnen mit Vektoren Da R eine vektorisierte Sprache ist, werden alle elementaren Rechenoperationen vektorisiert ausgeführt: ```r c(1,2,3) + c(1,1,1) # -> 2,3,4 c(1,2) * c(1,4) # -> 1,8 ``` Achtung, wenn Vektoren nicht gleich viele Elemente enthalten, dann wird das kürzere so oft wiederholt, bis es passt. Wenn es aufgeht, bekommt man nichts mit, ansonsten wird eine Warnung ausgegeben: ```r c(1,2,4) * 2 # 2,4,6 -> zweites Objekt wird umgewandelt in c(2,2,2) c(1,2,4) * c(2,3) # 2,6,8 -> mit Warnung ``` --- # 2. Objekte ### Indizierung von Vektoren Um einzelne Elemente von Vektoren anzusprechen, kann man Vektoren indizieren, z.B. vierter Eintrag des Vektors `x` wird durch `x[4]` indiziert. Folgende Möglichkeiten bestehen: - numerische Indizierung (auch mit Vektor): `x[c(2,3,7)]` indiziert das 2., 3. und 7. Element des Vektors `x`. - logische Indizierung: `x[c(TRUE, FALSE, TRUE, FALSE, FALSE)]` indiziert 1. und 3. Element aus `x` (wenn `x` aus 5 Elementen besteht) - Benannte Vektoren lassen sich auch über den Namen indizieren: `x["eins"]` indiziert das Element mit dem Namen "eins" - `x[-1]` indiziert alle Elemente auser dem ersten - `x[ x > 2 ]` indiziert alle Elemente, die größer als 2 sind (`x` sollte dazu numerisch sein) --- # 2. Objekte ### Nützliche Funktionen: <img src="figures/funktionen-vecs.png" style="width: 100%" align="center" /> --- # 2. Objekte ### Aufgabe 2.2 Schreiben Sie eine Funktion, die die Summe zweier Vektoren berechnet und als Wert (nicht als print-Ausgabe) den Character "Die Summe ist [Wert]" zurückgibt. Testen sie mit den Vektoren: ```r xy <- c(1,2,3) yx <- c(4,5,6) ``` Hinweis, sehen Sie sich dazu die Hilfe zur Funktion `paste()` an. --- # 2. Objekte ### Matrizen Mit der Funktion `matrix(data= NA, ncol=1, nrow =1, byrow = FALSE)` werden Matrizen erzeugt: ```r matrix(data = 1:9, ncol = 3, nrow = 3) # erst werden Spalten, dann Zeilen befüllt ``` ``` ## [,1] [,2] [,3] ## [1,] 1 4 7 ## [2,] 2 5 8 ## [3,] 3 6 9 ``` Ist `data` nicht lang genug, wird es entsprechend verlängert: ```r matrix(data = 1, ncol = 3, nrow = 3) # erst werden Spalten, dann Zeilen befüllt ``` ``` ## [,1] [,2] [,3] ## [1,] 1 1 1 ## [2,] 1 1 1 ## [3,] 1 1 1 ``` --- # 2. Objekte ### Matrizen ```r matrix(data = c(1,2), ncol = 3, nrow = 3) # erst werden Spalten, dann Zeilen befüllt ``` ``` ## Warning in matrix(data = c(1, 2), ncol = 3, nrow = 3): Datenlänge [2] ist kein ## Teiler oder Vielfaches der Anzahl der Zeilen [3] ``` ``` ## [,1] [,2] [,3] ## [1,] 1 2 1 ## [2,] 2 1 2 ## [3,] 1 2 1 ``` Entwicklung nach Zeilen: ```r matrix(data = 1:9, ncol = 3, nrow = 3, byrow = TRUE) ``` ``` ## [,1] [,2] [,3] ## [1,] 1 2 3 ## [2,] 4 5 6 ## [3,] 7 8 9 ``` --- # 2. Objekte ### Matrizen - Indizierung Die Indizierung ist analog zur Indizierung von Vektoren (also numerisch, logisch, mit Namen,...) allerdings benötigen wir einen Zeilen- und Zeilenindex: ```r x <- matrix(data = 1:9, ncol = 3, nrow = 3, byrow = TRUE) x[1,3] # Element, das in der 1. Zeile und 3. Spalte steht x[1,] # gibt erste Zeile zurück (als Vektor) x[,2] # gibt zweite Spalte zurück (als Vektor) x[,2, drop = FALSE] # gibt zweite Spalte zurück (als 3x1-Matrix) x[x>2] # Elemente, deren Wert größer als 2 ist (als Vektor) ``` - Mit `cbind()`und `rbind()` lassen sich Matrizen spaltenweise bzw. zeilenweise "stacken" - `dim()` gibt die Anzahl der Elemente in jeder Dimension an --- # 2. Objekte ### Nützliche Funktionen für Matrizen: <img src="figures/functions-mat.png" style="width: 100%" align="center" /> --- # 2. Objekte ### Arrays Matrizen haben zwei Dimensionen, wie wir gesehen haben. Eine Verallgemeinerung sind Arrays mit beliebig vielen Dimensionen. Diese werden mit der Funktion `array(data, dim)` erzeugt. Dabei ist `dim` ein Vektor, der die Anzahl der Elemente in jeder Dimensionen angibt. ```r array(1:30, dim = c(3,3,5)) ``` ist ein Array das die Dimension `\(3 \times 3 \times 5\)` hat. - Indizierung analog zu der von Matrizen - elementare Rechenoperationen werden alle elementweise ausgeführt: ```r A <- matrix(1:6, nrow = 2) A^2 ``` ``` ## [,1] [,2] [,3] ## [1,] 1 9 25 ## [2,] 4 16 36 ``` --- # 2. Objekte ### Aufgabe 2.3 Berechnen Sie für die Matrix `A = Mat_A` `Mat_A <- matrix(1:9, ncol = 3)` und den Vektor `b = vec_b` `vec_b <- 12:14` das Matrizenprodukt `\(A\cdot b\)` und das Komponentenprodukt. Erklären Sie die Unterschiede. --- # 2. Objekte ### Aufgabe 2.4 Geben Sie die Objekte `\(y = (3,5,2,8,6,4,7)'\)` und die Matrix `\(X\)`, deren erste Spalte aus Einsen besteht und deren zweite Spalte die Einträge `\((4,3,7,1,3,7,5)'\)` enthält, in R ein. 1. Geben Sie für `X` und `y` jeweils die 3. Beobachtung dieses Regressionsmodells aus. 2. Berechnen Sie `\(X'X\)`, `\(X'y\)`, `\((X'X)^{-1}\)` sowie den KQ-Schätzer des linearen Regressionsmodells.<br> **Hinweis**: die Formel für den KQ-Schätzer ist `\(\hat{\beta} = (X'X)^{-1}X'y\)`. 3. Geben Sie unter Standardannahmen den Standardfehler der Koeffizienten an.<br> **Hinweis**: Falls Sie die Definition nicht mehr wissen, sehen Sie im Skript zu [Ökonometrie 1](https://www.uni-regensburg.de/wirtschaftswissenschaften/vwl-tschernig/medien/zeitreihenoekonometrie/eoe_ws19_chapter_all.pdf) nach. 4. Erzeugen Sie eine Funktion, die bei Angabe von `y` und `X` eine Matrix mit der KQ-Schätzung und den Standardfehlern als Spalten ausgibt. --- # 2. Objekte ### Aufgabe 2.5 Geben Sie die Matrix `$$A = \begin{pmatrix} 1.0 & 0.5\\ 0.5 & 1.0 \end{pmatrix}$$` in R ein. Berechnen Sie die Eigenwertzerlegung `\(V\cdot \Lambda\cdot V^{-1}\)` von `\(A\)`, wobei `\(\Lambda\)` eine Diagonalmatrix mit den Eigenwerten und `\(V\)` die Matrix mit den Eigenvektoren als Spalten ist. Ist obiges Produkt gleich `\(A\)`? --- # 2. Objekte ### Listen Bei Vektoren, Matrizen und Arrays mussten alle Elemente gleichen Datentyps sein. Listen sind dahingehend allgemeiner, als dass sie beliebige Kombinationen von Datentypen erlauben. Erzeugt wird eine Liste mit der Funktion `list()`. ```r L1 <- list( a = 1:3, A = matrix(1:9,3,3), w = "Hallo!" # Elemente können Namen haben UND beliebiger Art sein! ) ``` Listen können wieder Listen enthalen: ```r L2 <- list( a = 1:3, l1 = L1 # Liste in Liste ) ``` --- # 2. Objekte ### Listen Die Indizierung von Listen erfolgt mit zwei eckigen Klammern (numerisch, logisch, mit Namen): ```r L1[[1]] # 1,2,3 -> Vektor L1[1] # list(1:3) -> noch eine Liste (Teilliste von L1) L1[["w"]] # "Hallo!" ``` Benannte Listen lassen sich auch mit dem `$`-Operator indizieren: ```r L1$w # "Hallo!" ``` --- # 2. Objekte ### Aufgabe 2.6 - Erzeugen Sie selbst eine Liste mit drei verschiedenen Datentypen und Namen. - Benutzen Sie selbst eine numerische Indizierung und eine Indizierung per Namen. - Benutzen Sie außerdem die `str()`-Funktion auf Ihre Liste. - Machen Sie nun eine Liste von Listen von Listen. --- # 2. Objekte ### Dataframes Dataframes sind Listen, deren Elemente Vektoren gleicher Länge aber nicht notwendigerweise gleichen Datentyps sind. Typischerweise werden Datensätze in R in Dataframes gespeichert. Dataframes werden mit `data.frame()` erzeugt und können sowohl wie Matrizen als auch wie Listen indiziert werden. Beispiele: ```r Personen <- data.frame( Vorname = c("Patrick","Mario","Claudio","Mario"), Nachname = c("Meyer","Schröder","Müller","Schmidt"), GebDat = c("1994-03-03","1986-05-21","1978-10-03","1985-07-10"), Alter = c(25,33,41,34), AnzKinder = c(2,1,0,1), stringsAsFactors = FALSE # Namen sollen hier als "character" # eingelesen werden und nicht als "factor" ) ``` --- # 2. Objekte ### Dataframes Indizierung: ```r names(Personen) # Variablennamen Personen$Vorname # Indizierung wie bei Liste ($) Personen[ ,1] # Numerische Indizierung wie bei Matrix Personen[ ,"Vorname"] # Namentliche Indizierung Personen[Personen$Nachname=="Müller" , ] # logische Indizierung ``` Um Variablen zum Datensatz hinzuzufügen: ```r Personen$Geschlecht <- c("m", "m", "m", "m") ``` --- # 2. Objekte ### Dataframes Häufig möchte man aus Datensätzen bestimmte Beobachtunge selektieren, dazu gibt es die Funktion `subset()` und hilfreich ist dabei der Operator `%in%`: ```r subset(Personen, Vorname %in% c("Müller", "Schmidt")) ``` Wir werden aber später noch Funktionen aus dem R-Paket `dplyr` kennen lernen, womit sich solche Selektionen sehr flexibel gestalten lassen. Weitere hilfreiche Funktionen für `data.frames`: - `summary()`: gibt zusammenfassende Statistiken für einzelne Variablen aus - `head()`: zeigt ersten Zeilen an - `tail()`: zeigt die letzten Zeilen an - `attach()`: macht die Variablen des Datensatzes im globalen Workspace sichtbar - `split()`: teilt den Datensatz in einzelne Teildatensätze auf - `merge()`: vereinigt zwei Datensätze mit unterschiedlichen Variablen --- # 2. Objekte ### Aufgabe 2.7 Benutzen sie den gerade erstellten data.frame "Personen" und machen Sie folgendes: 1. Ändern Sie den Spaltennamen "Vorname" in "prename". 2. Sprechen Sie die Geburtsdaten auf zwei verschiedene Arten an (vgl. data.frame-Matrix-Listen-Ähnlichkeit). 3. Geben Sie sich eine logischen Indizierung aller Personen aus, die mehr als 2 Kinder haben. 4. Fügen Sie eine neue Beobachtung (Zeile) (z. B. Sie selbst) mit willkürlichen Daten hinzu. 5. Fügen Sie eine neue Eigenschaft (Spalte) der Personen hinzu. 6. Wählen Sie alle Personen, die mehr als einem Kind haben und jünger als 32 Jahre sind, aus. --- # 2. Objekte ### Weitere Objekte (Faktorvariable, Geordnete Kategoriale, Zeitreihe, Zeit-/Datumsangaben) Diese speziellen Objekte sind keine atomaren Datentypen. Sie sind aber in vielen Anwendungen hilfreich. ## Faktorvariablen Häufig liegt Information zu Gruppen vor (kategoriales Merkmal), Beispiele: - Geschlecht - Automarke - Wochentag - Bundesland für diese Art von Information gibt es in R Faktorvariablen, diese werden mit `factor()` erzeugt: ```r x <- rep(c(1,2), 4) factor(x, labels=c("weiblich", "männlich")) Z <- c("Ja","Ja","Nein","Ja","Vielleicht","Nein","Vielleicht","Ja","Ja","Nein") factor(Z) ``` --- # 2. Objekte ### Warum Faktoren - weniger Speicherplatz - in Plots und Regressionen verwendbar (vgl. coplots später) - `summary()`, `plot()` stellen entsprechende Funktionalität bereit: ```r x <- sample(x=1:2, size=100, replace = TRUE) x <- factor(x, labels = c("männlich", "weiblich")) plot(x) ``` <!-- --> ```r summary(x) ``` ``` ## männlich weiblich ## 57 43 ``` --- # 2. Objekte ### Geordnete Kategorien Häufig liegen auch kategoriale Daten vor, die sinnvoll geordnet werden können. ##### Beispiel: In Surveys werden werden häufig Fragen gestellt wie *"Bier ist das wichtigste Getränk der Welt!"* mit den Antwortmöglichkeiten: - `stimme ich absolut zu` - `stimme ich teilweise zu` - `teils/teils` - `stimme eher nicht zu` - `stimme überhaupt nicht zu` Hier ist eine Ordnung sinnvoll und diese Information kann in einigen statistischen Verfahren nützlich sein. --- # 2. Objekte ### Geordnete Kategorien Erzeugt werden solche geordneten Faktorvariablen mit der Funktion `ordered()`: ```r ord <- c("sehr gut","mittel","gut","schlecht","gut","schlecht","mittel","sehr gut","gut") class(ord) # character OF <- ordered( ord , levels=c("sehr schlecht","schlecht","mittel","gut","sehr gut")) class(OF) # ordered, factor ``` ### Aufgabe 2.8 Erzeugen Sie einen Faktor mit den Ausprägungen `"Hessen", "Bayern", "Baden-Württemberg"` und `"Thüringen"`. Erstellen sie auch einen geordneten Faktor, sortiert nach alphabetischer Reihenfolge. --- # 2. Objekte ### Datums- und Zeitangaben Manchmal braucht man Uhrzeiten und/oder Datumsangaben. Hierfür existieren eigene Datenstrukturen: ```r Sys.time() # aktuelle Systemzeit, z.B. "2019-11-08 19:03:43 CET" class(Sys.time()) # "POSIXct" "POSIXt" ?POSIXct # Hilfe zu DateTimeClasses ``` Die Klasse `POSIXct` ist eine Datenstruktur, die eine Zeitangabe als Anzahl der Sekunden seit dem 1. Januar 1970 00:00:00 UTC speichert: ```r x <- as.POSIXct("2019-11-24 10:12:05", tz = "UTC") # mit Übergabe der 'timezone' as.numeric(x) # -> 1574590325 ``` --- # 2. Objekte ### Datums- und Zeitangaben POSIX Zeitangaben sind eine ziemlich verbreitete Konvention um in Computern Zeiten darzustellen. In R gibt es eine Vielzahl an Darstellungsmöglichkeiten der Klasse `POSIXct`/`POSIXlt` über die zugehörige `format()`-Methode (siehe dazu auch die Hilfe zur Funktion `strptime()` und Eintrag auf [R-Bloggers](https://www.r-bloggers.com/date-formats-in-r/)): kleine Auswahl: - `%m`: Monat als Dezimalzahl (01-12) - `%y`: Jahr ohne Jahrhundertangabe (00 - 99) - `%Y`: Jahr mit Jahrhundertangabe z.B. 1987 - `%H`: Stunde als Dezimalzahl Beispiel: ```r x <- as.POSIXct("2019-11-24 10:12:05", tz = "UTC") format(x, format = "%Y-%m: %H Uhr") # "2019-11: 10 Uhr" ``` --- # 2. Objekte ### Datums- und Zeitangaben Häufig ist die Uhrzeit nicht nötig sonder nur das Datum, dafür existiert Klasse `Date`. ```r x <- as.Date("2019-11-24") # Standardformat: %Y-%m-%d class(x) # "Date" x <- as.Date("24.11.2019", format = "%d.%m.%Y") # anderes Format muss angegeben werden as.numeric(x) # 18224 (Anzahl der Tage seit 1.1.1970) ``` Mit `Date`- und `POSIXct`-Klasse lässt sich auch rechnen: ```r (x <- as.Date("2019-11-24") - as.Date("2019-11-14")) # -> Time difference of 10 days class(x) # "difftime" ``` --- # 2. Objekte ### Aufgabe 2.9 Heute ist der 15.11.2019. 1. Lesen Sie dieses Datum ein 2. Formatieren Sie es in R als: - "20191115" - "2019-Nov" - "Kalenderwoche: 45" ### Aufgabe 2.10 Am 27.10.2013 war eine Zeitumstellung von Sommerzeit auf Winterzeit. Versuchen Sie diese Stunde in R zu reproduzieren. <br> **Hinweis**: Es reicht zu zeigen, dass dieser Tag 25 Stunden hatte. --- # 2. Objekte ### Zeitreihendaten Für Zeitreihen gibt es mehrere mögliche Klassen, z.B.: `ts` (aus dem Paket `stats`)und `zoo` (bereitgestellt vom Pakte `zoo`): ```r ald <- ts( data = c( 4284691,4247561,4124836,3976555, 3812335,3687597,3715509,3705949, 3543866,3434067,3378747,3406389, 3659316,3617418,3507383,3413881), start = c(2007,1), frequency = 12) # Startet im Januar 2007, monatliche Zeitreihe class(ald) # "ts" ``` `ts`-Objekte können nur regelmäßige Zeitreihen sein. Das ist manchmal unpraktisch (z.B. Handelstage Börse). Für unregelmäßige Zeitreihen gibt es z.B. `zoo`: ```r a <- 1:10 a.Date <- seq( from = as.Date("2010-01-01"), to = as.Date("2020-01-01"), by = "years")[-3] a.zoo <- zoo(x = a, order.by = a.Date) ``` --- # 2. Objekte ### Aufgabe 2.11 Ändern Sie im Datensatz `Personen` aus [Aufgabe 2.7](#59) die Variable `GebDat` in eine Datumsvariable um. Fügen Sie anschließend dem Datensatz eine weitere Variable `AlterTage` hinzu, welche das Alter der Personen in Tagen angibt. --- # 2. Objekte ### Daten einlesen Um Daten in R einzulesen gibt bestehen verschiedene Möglichkeiten, je nachdem, in welcher Form die Daten vorliegen: - Liegen Daten als R-Image (`.RData`) vor, lassen sich die darin erhaltenen Objekte einfach mit `load()` in den Workspace laden. - Häufig werden Datensätze als strukturierte Textdateien zur Verfügung gestellt, z.B. der Datensatz `Personen` sieht als `csv`-Datei (mit `;` als Spaltentrenner) folgendermaßen aus: ```c "Vorname";"Nachname";"GebDat";"Alter";"AnzKinder" "Patrick";"Meyer";"1994-03-03";25;2 "Mario";"Schröder";"1986-05-21";33;1 "Claudio";"Müller";"1978-10-03";41;0 "Mario";"Schmidt";"1985-07-10";34;1 ``` Zum Einlesen strukturierter Texte gibt es die Funktionen `read.table()`, `read.csv()`, `read.csv2()`, `read.delim()` und `read.delim2()`, jeweils mit anderen default-Belegungen. --- # 2. Objekte ### Daten einlesen - `xls`/`xlsx`-Dateien: eine ganze Weile stand R mit Excel-Dateien auf Kriegsfuß, inzwischen gibt es aber das Paket `readxl`, welches die Funktionen `read_xls()`und `read_xlsx()` bereitstellt. - für Daten von anderen Statistikpaketen (Stata, SPSS, Eviews, SAS,...) gibt es die Pakete `foreign` und `haven`, welche entsprechende Funktionen für die jeweiligen Dateiformate bereitstellen. Beispiele: ```r data_pers <- read.table("personen.csv", header = TRUE, ## Variablennamen in erster Zeile sep = ";" ## Spaltentrenner ";" ) ``` --- # 2. Objekte ### Daten einlesen Weitere Beispiele (dazu erstmal zip-Ordner mit Beispieldateien runterladen) ```r ## herunterladen der Beispieldateien curl::curl_download(url = "http://pc1011601230.ur.de/example_data.zip", destfile = "example_data.zip") ## zip entpacken res <- unzip("example_data.zip") ``` ```r install.packages("readxl", "haven") ## pakete installieren falls nicht lokal vorhanden library(readxl) excel_data <- read_xls("Africa.xls", skip = 7, col_names = c("country","pop", "larea","pop_dens", "gdpppp", "gdppcppp","gdpgrwth")) dax <- read.csv("dax.csv") ## default werte für sep, dec, header passen schon! schools_treat <- haven::read_dta("TreatmentSchools.dta") ## Angabe Namensraum über "::" ``` --- # 2. Objekte ### Aufgabe 2.12 Lesen Sie die Arbeitslosenzahlen (aus dem Jahr 2013) in Deutschland (Anzahl, Quote) auf Kreisebene in R ein. Laden Sie dazu die entsprechende [Excel-Tabelle](https://www.uni-regensburg.de/wirtschaftswissenschaften/vwl-tschernig/medien/programmieren-mit-r/alq_kreisebene.xla) von der Kurshompage herunter.<br> Wandeln Sie den relevanten Teil der xls-Tabelle in ein Komma- oder Tab-Getrenntes Format um. Verwenden Sie die Funktion `read.table` und lesen Sie den Datensatz als `data_arge` ein. Nach dem Einlesen machen Sie folgendes: - Summary ausgeben lassen (Überprüfen, ob alle Variablen passen) - Falls Sie die Überschriften nicht eingelesen haben, übergeben Sie dem Datensatz nun folgende Überschriften: `Kreisschlüssel`, `Kreis`, `Abs_tot`,`Quote_tot`, `Quote_AbhEP`. - Nutzen Sie die Teilmenge des Datensatzes, die nur noch Kreis, Abs_tot, Quote_tot beinhaltet. --- # 2. Objekte ### Aufgabe 2.12 (Fortsetzung) - Berechnen Sie von dieser Teilmenge ausgehend die gesamte Erwerbsbevölkerung und die gesamte Arbeitslosenquote - Trunkiert man den Kreisschlüssel, nachdem man ihn durch 1000 teilt, so gehört jede Zahl 1 bis 16 zu einem Bundesland. Die Reihlenfolge ist dabei:<br> `"Schleswig-Holstein", "Hamburg", "Niedersachsen", "Bremen", "NRW", "Hessen", "Rheinland-Pfalz", "BW", "Bayern", "Saarland", "Berlin", "Brandenburg", "Mecklenburg-Vopo", "Sachsen", "Sachsen-Anhalt", "Thüringen"`<br> Fügen Sie nun eine neue Spalte ein, die zu jeder Beobachtung angibt, in welchem Bundesland sie ist. <br> **Hinweis**: Sie könnten die Funktion `trunc()` gebrauchen. - Benutzen Sie nun den `order()` Befehl und geben Sie sich die Kreise mit den 30 höchsten Arbeitslosenquoten aus. - Speichern Sie nun den Datensatz als `ALQ_Kreise.txt` ab und den (kompletten) data.frame inklusive der Bundesländer als `alq.RData`. (Letzter samt der Bundesländer wird später noch benötigt) --- # 2. Objects ### Noch mehr Input/Output - meist können Daten genauso wie sie eingelesen werden auch wieder auf der Festplatte gespeichert werden: `write.csv()`, `write.table()`, `haven::write_dta()`,... - Es gibt weitere, elementarere Möglichkeiten, um Daten zu lesen oder zu schreiben: `readLines()`/`writeLines()`, `scan()`/`write()`,... - Anbindungen an Datenbanken mit dem `odbc`-Package - Möglichkeit, aus der Zwischenablage zu lesen nachdem [Personen](#70) kopiert wurde: ```r personen <- read.table(file(description = "clipboard"), sep = ";") ``` - Netzwerkressourcen über `url()`, komprimierte Dateien mit `unz()`,... Siehe dazu auch `?connections()` --- # 3. Grafiken - In diesem Kapitel behandeln wir Grafikfunktionen aus dem `graphics`-Paket - Es gibt auch andere Grafik-Pakete, z.B. `ggplot2` (siehe Dazu [Buch](https://ggplot2-book.org/) von Hadley Wickham), `plotly`, `Rgnuplot`,... Zum Erzeugen von Plots mit den R Standard `graphics`-Paket existieren sogenannte *high-level*- und *low-level*-Plot-Funktionen. - *high-level*-Funktionen erzeugen eine eigene Grafik (und öffnen ein *device*) - *low-level*-Funktionen fügen einer bereits bestehenden Grafik weitere Elemente hinzu Einige *devices* gibt es? Auswahl: - `windows()`/`x11()`/`quartz()`: Bildschirmgrafiken (windows, Unix, Mac), (wird default) - `pdf()`: Adobe PDF (leicht in LaTeX einzubinden) - `svg()`: Scalable Vector Graphics (Häufig in Webseiten genutzt) - `png()`, `jpeg()`, `tiff()`, `bmp()`: unterschiedliche Bitmaps-Formate --- # 3. Grafiken Grafik erzeugen: ```r pdf("myPlot.pdf") ## plot-device öffnen plot(y = rnorm(100), x = 1:100) ## Grafik erzeugen, mit high-level funktion plot() dev.off() ## plot-device schließen ``` `plot()` ist eine Funktion, die unterschiedliche Methoden für unterschiedliche Objekte aufruft: ```r function (x, y, ...) UseMethod("plot") <bytecode: 0x55ef0a3b5ec8> <environment: namespace:graphics> ``` Diese Methoden sind die unterschiedlichen zur Verfügung stehenden high-level-Plotfunktionen --- # 3. Grafiken ### Arten von Plots (High-Level Plots) - `barplot()`: Säulen- bzw. Balkendiagramm - `pie()`: Kuchendiagramm (package plotrix mit pie3d) - `boxplot()`: Boxplot - `contour()`: Gut geeignet für Höhenkarten, d.h. f: R^2 -> R; filled.contour für farbibe Höhenkarten - `coplot()`: conditioning plot: Für einen factor mehrfach ausgegebene Plots - `curve()`: Linienplot, aber einfache Funktionsübergabe durch curve(f(x)= ..., x=, from=, to) - `dotchart()`: auch mit Scatterplot erzeugbar; besonders sinnvoll für sehr viele Faktoren mit einer Ausprägung - `hist()`: Histogramm - `mosaicplot()`: Mosaikplot; sinnvoll für Zusammensetzungen in der Zeit - `pairs()`: Sinnvoll zur Korrelationsüberprüfung - `image()`: Zum Zeichnen... - `persp()`: Wieder zum Zeichen von 3d Graphen, inklusive Koordinatenkreuzwahl - `scatterplot3d()`: das gleiche - `qqplot()`: Quantile-Quantile-Plot --- # 3. Grafiken Beispiele: ```r load(url("http://pc1011601230.ur.de/CPS1985.RData")) class(CPS1985) ``` ``` ## [1] "data.frame" ``` ```r plot(CPS1985) ## plot auf data.frame ruft pairs() auf ``` <!-- --> --- # 3. Grafiken Beispiele: ```r class(CPS1985$wage) ``` ``` ## [1] "numeric" ``` ```r plot(CPS1985$wage) ## plot auf numeric ruft plot.default() auf ``` <!-- --> --- # 3. Grafiken Beispiele: ```r class(CPS1985$occupation) ``` ``` ## [1] "factor" ``` ```r plot(CPS1985$occupation) ## plot auf factor ruft barplot() auf ``` <!-- --> --- # 3. Grafiken ### Eine stetige Variable ```r hist(CPS1985$wage , breaks = 20) plot(density(CPS1985$wage)) ``` <!-- --> --- # 3. Grafiken ### Eine diskrete Variable ```r plot(CPS1985$occupation) ## erzeugt barplot(table(CPS1985$occupation)) pie(table(CPS1985$occupation)) ## gleiche Information, aber nicht unbedingt zu empfehlen ``` <!-- --> --- # 3. Grafiken ### Zwei stetige Variablen ```r ## Scatterplot als Ausgangspunkt der meisten Überlegungen: plot(x = CPS1985$education, y = CPS1985$wage) ``` <!-- --> --- # 3. Grafiken ### Zwei diskrete Variablen ```r ## Scatterplot als Ausgangspunkt der meisten Überlegungen: plot(gender ~ occupation, data = CPS1985) ``` <!-- --> --- # 3. Grafiken ### Stetige Variable in Abhängigkeit von einer diskreten Variable ```r plot(wage ~ occupation, data = CPS1985) ``` <!-- --> --- # 3. Grafiken ### drei oder mehr Variablen ```r ## nacheinander plot(wage ~ education + occupation, data = CPS1985) ``` <!-- --> --- # 3. Grafiken ```r ## in einem plot coplot(wage ~ education | occupation, data = CPS1985) ``` <!-- --> --- # 3. Grafiken ```r ## vier variablen in einem plot coplot(wage ~ education | occupation + gender, data = CPS1985) ``` <!-- --> --- # 3. Grafiken ### Grafiken anpassen ```r hist(CPS1985$education, breaks = 3) ## wie 'fein' soll das Histogramm sein hist(CPS1985$education, breaks = 16) ``` <!-- --> --- # 3. Grafiken ```r plot(CPS1985$wage, type = "p") ## points plot(CPS1985$wage, type = "l") ## lines ``` <!-- --> --- # 3. Grafiken ```r # Überschrift und Achsenbeschriftung with(CPS1985,{ ## siehe Hilfe zu with() plot(x = age, y = wage, main = "Streudiagramm von Alter und Stundenlohn") plot(age, wage, xlab = "Alter in Jahren", ylab = "Stundenlohn", main = "Streudiagramm von Alter und Stundenlohn") }) ``` <!-- --> --- # 3. Grafiken ### Feintuning Nicht alles lässt sich über optionale Argumente in high-level-Funktionen anpassen, dann häufig über `par()`: .pull-left[ ```r par(bg = "yellow") with(CPS1985, plot(age) ) ``` <!-- --> ] .pull-right[ ```r par(bg = "transparent", cex=0.5) with(CPS1985, plot(age) ) ``` <!-- --> ] --- # 3. Grafiken **Übersicht**: Häufig genutzte Argumente von Grafikfunktionen - `axes`: Achsenangabe - `bg`: Backgroundfarbe - `cex`: Faktor, der die Vergrößerung zum Standard angeben soll - `col`: Plotfarbe - `log`: xlog und ylog für logarithmische Skalen - `lty`, `lwd`: Linientyp und -dicke - `mai`: 4-Vektor über die Ränder unten, links, oben, rechts - `main`, `sub`: Titel, Untertitel - `mar`: Rand - `mfcol`, `mfrow`: mehrere Plots in ein Grafikfenster (spaltenweise/zeilenweise) - `pch`: Pointcharakter (1-16) - `usr`: Die Extremalstellen für einen plot - `xlab`, `ylab`: x und y Beschriftung - `xlim`, `ylim`: x und y Begrenzung Siehe `?par` für alle Grafikoptionen --- # 3. Grafiken ### Low-level Grafik-Funktionen - `lines`: Linien einzeichnen - `abline`: Schnell für horizontale, vertikale Geraden und Geraden in Geradengleichung `\(y = bx+a\)` - `points`: Punkte - `arrows`: Pfeile - `polygon`: Beliebige Vielecke - `segments`: Unausgemalte Vielecke - `axis`: Achsen - `grid`: Gitter - `rug`: "Dichteteppich" - `title`: Titel - `legend`: Legende - `text`: Text durch `\((x,y)\)` Koordinaten - `mtext`: Text durch Positionsangebae wie `side=1,...,4` --- # 3. Grafiken .pull-left[ ```r attach(CPS1985) ## siehe ?attach *plot(x = education, y = wage, * pch = 16, axes = FALSE) ``` ] .pull-right[ <!-- --> ] --- # 3. Grafiken .pull-left[ ```r attach(CPS1985) ## siehe ?attach plot(x = education, y = wage, pch = 16, axes = FALSE) # Achsen hinzufügen *ticks <- seq(-100, 100, 0.5) *axis(side = 1, at = ticks, * labels = paste(ticks,"y")) *axis(side = 2, at = seq(-100,100, 0.5)) ``` ] .pull-right[ <!-- --> ] --- # 3. Grafiken .pull-left[ ```r attach(CPS1985) ## siehe ?attach plot(x = education, y = wage, pch = 16, axes = FALSE) # Achsen hinzufügen ticks <- seq(-100, 100, 0.5) axis(side = 1, at = ticks, labels = paste(ticks,"y")) axis(side = 2, at = seq(-100,100, 0.5)) # Striche wo Datenpunkte *rug(wage, side = 2) ``` ] .pull-right[ <!-- --> ] --- # 3. Grafiken .pull-left[ ```r attach(CPS1985) ## siehe ?attach plot(x = education, y = wage, pch = 16, axes = FALSE) # Achsen hinzufügen ticks <- seq(-100, 100, 0.5) axis(side = 1, at = ticks, labels = paste(ticks,"y")) axis(side = 2, at = seq(-100,100, 0.5)) # Striche wo Datenpunkte rug(wage, side = 2) # Achsenabschnitt und Steigung angeben *abline(a = -0.7460, b=0.7505, col="blue") *abline(h = mean(wage), lty=2, * col = "red", lwd = 8) *abline(v = mean(education), lty=3, * col = "orange", lwd = 4) ``` ] .pull-right[ <!-- --> ] --- # 3. Grafiken .pull-left[ ```r plot(x = education, y = wage, pch = 16) ``` ] .pull-right[ <!-- --> ] --- # 3. Grafiken .pull-left[ ```r plot(x = education, y = wage, pch = 16) *lines(x = c(2, 7, 10), * y = c(20, 30, 25), * col="darkgreen", lwd = 3) ``` ] .pull-right[ <!-- --> ] --- # 3. Grafiken .pull-left[ ```r plot(x = education, y = wage, pch = 16) lines(x = c(2, 7, 10), y = c(20, 30, 25), col="darkgreen", lwd = 3) *points(x = c(2, 7, 10), * y = c(20, 30, 25), * col="darkred", pch = 19) ``` ] .pull-right[ <!-- --> ] --- # 3. Grafiken .pull-left[ ```r plot(x = education, y = wage, pch = 16) lines(x = c(2, 7, 10), y = c(20, 30, 25), col="darkgreen", lwd = 3) points(x = c(2, 7, 10), y = c(20, 30, 25), col="darkred", pch = 19) *polygon(x = c(3, 3, 10, 8), * y = c(10,20,20,10), * col="lightgreen") ``` ] .pull-right[ <!-- --> ] --- # 3. Grafiken .pull-left[ ```r plot(x = education, y = wage, pch = 16) lines(x = c(2, 7, 10), y = c(20, 30, 25), col="darkgreen", lwd = 3) points(x = c(2, 7, 10), y = c(20, 30, 25), col="darkred", pch = 19) polygon(x = c(3, 3, 10, 8), y = c(10,20,20,10), col="lightgreen") *arrows(x0 = 10, y0 = 40, x1 = 13.8, * y1 = 44.2, lwd = 5, * col = "darkblue") ``` ] .pull-right[ <!-- --> ] --- # 3. Grafiken .pull-left[ ```r plot(x = education, y = wage, pch = 16) lines(x = c(2, 7, 10), y = c(20, 30, 25), col="darkgreen", lwd = 3) points(x = c(2, 7, 10), y = c(20, 30, 25), col="darkred", pch = 19) polygon(x = c(3, 3, 10, 8), y = c(10,20,20,10), col="lightgreen") arrows(x0 = 10, y0 = 40, x1 = 13.8, y1 = 44.2, lwd = 5, col = "darkblue") *title("Lohn versus Ausbildung") ``` ] .pull-right[ <!-- --> ] --- # 3. Grafiken .pull-left[ ```r plot(x = education, y = wage, pch = 16) lines(x = c(2, 7, 10), y = c(20, 30, 25), col="darkgreen", lwd = 3) points(x = c(2, 7, 10), y = c(20, 30, 25), col="darkred", pch = 19) polygon(x = c(3, 3, 10, 8), y = c(10,20,20,10), col="lightgreen") arrows(x0 = 10, y0 = 40, x1 = 13.8, y1 = 44.2, lwd = 5, col = "darkblue") title("Lohn versus Ausbildung") *text(x = 11, y = 30, "Text im Plot") ``` ] .pull-right[ <!-- --> ] --- # 3. Grafiken .pull-left[ ```r plot(x = education, y = wage, pch = 16) lines(x = c(2, 7, 10), y = c(20, 30, 25), col="darkgreen", lwd = 3) points(x = c(2, 7, 10), y = c(20, 30, 25), col="darkred", pch = 19) polygon(x = c(3, 3, 10, 8), y = c(10,20,20,10), col="lightgreen") arrows(x0 = 10, y0 = 40, x1 = 13.8, y1 = 44.2, lwd = 5, col = "darkblue") title("Lohn versus Ausbildung") text(x = 11, y = 30, "Text im Plot") *legend(x="topright", * legend = c("Datenpunkte","Linie"), * lty = c(NA, 1), pch = c(19, NA), * lwd = c(1, 3), col = c(1, "darkgreen")) ``` ] .pull-right[ 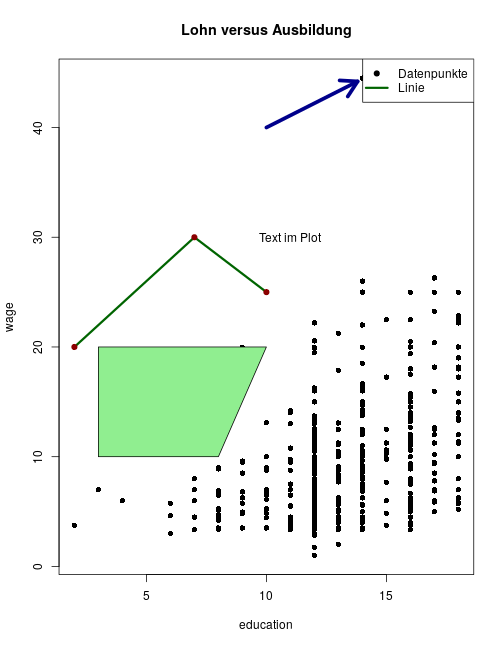<!-- --> ] --- # 3. Grafiken ### Aufgabe 3.1 Zeichnen Sie einen Plot der Funktion `\(f(x)=e^x+2\)` im Bereich von -2 bis +2, Linienfarbe grün Erstellen Sie eine Legende oben links mit dem Eintrag `\(e^2\)` und der Linie. **Hinweis**: Um mathematische Beschriftungen in eine Legende zu machen, benötigen Sie die "mathematical annotations", die [hier](http://stuff.mit.edu/afs/sipb/project/r-project/arch/i386_rhel3/lib/R/library/graphics/html/plotmath.html) zu finden sind. Mit `paste()` und `expression()` dürfte es danach kein Problem mehr sein. ### Aufgabe 3.2 Erzeugen Sie einen Scatterplot von `wage` gegen `education` (aus dem Datensatz `CPS1985`), bei dem Frauen als rote und Männer als blaue Punkte erscheinen. Erzeugen Sie eine Legende, die dies illustriert. ### Aufgabe 3.3 Versuchen Sie einen Christbaum zu zeichnen (z. B. mit `polygon()`). Speicheren Sie diesen als `.pdf` im "graphs"-Ordner ab. --- # 4. Datenanalyse In diesem Kapitel werden wir uns (oberflächlich) mir dem Paket [dplyr](https://dplyr.tidyverse.org/) befassen. Dieses Paket ist Teil des [tidyverse](https://www.tidyverse.org/), einer Sammlung von R-Paketen, die dazu entwickelt wurde, die Arbeit mit Daten möglichst einheitlich zu gestalten. Folgende Pakete gehören zum [tidyverse](https://www.tidyverse.org/): - [dplyr](https://dplyr.tidyverse.org/): "Grammar of Data Manipulation" - [ggplot2](https://ggplot2.tidyverse.org/): "Grammar of Graphics" - [readr](https://readr.tidyverse.org/): "fast and friendly way to read rectangular data" - [tibble](https://tibble.tidyverse.org/): "A tibble, or tbl_df, is a modern reimagining of the data.frame" - [tidyr](https://readr.tidyverse.org/): "create tidy data. Tidy data is data where: 1. Every column is [a] variable. 2. Every row is an observation.. 3. Every cell is a single value." - [purrr](https://purrr.tidyverse.org/): "enhance R’s functional programming toolkit" --- # 4. Datenanalyse Ein wichtiger Baustein bei der Arbeit mit Daten und `dplyr` ist der im Paket `magrittr` enhaltene Pipe-Operator `%>%`. Ziel dieses (in vielen anderen Sprachen ebenfalls vorhandenen) Operators ist es, Funktionenkomposition möglichst einfach lesbar im Code darzustellen. Beispiel: ```r library(dplyr) f <- function(x) x + 10 g <- function(x) x * 2 a <- 2 f(g(a)) # 2*a + 10 -> 14 ## das gleiche Ergebnis mit dem Pipe-Operator a %>% g %>% f ``` --- # 4. Datenanalyse Weitere Beispiele .pull-left[ #### dplyr mit `%>%` ```r Personen %>% select(Alter) %>% summary() CPS1985 %>% filter(age > 25) %>% group_by(gender) %>% summarise(mwage = mean(wage)) ``` ] .pull-right[ #### standard R ```r summary( subset(Personen, select = Alter) ) datatemp <- subset(CPS1985, age > 25) tapply(X = datatemp$wage, INDEX = datatemp$gender, FUN = mean) ``` ] Im zweiten Beispiel sieht man bereits, dass man sich mit dem Pipe-Operator häufig nicht benötigte Zuweisungen sparen kann --- # 4. Datenanalyse #### `dplyr` Die wesentlichen Funktionen von `dplyr` sind: - [`mutate()`](https://dplyr.tidyverse.org/reference/mutate.html): neue Variablen einem Datensatz hinzufügen - [`select()`](https://dplyr.tidyverse.org/reference/select.html): Variablen (Spalten) auswählen - [`filter()`](https://dplyr.tidyverse.org/reference/filter.html): Beobachtungen (Zeilen) auswählen - [`arrange()`](https://dplyr.tidyverse.org/reference/arrange.html): Beobachtungen sortieren - [`summarise()`](https://dplyr.tidyverse.org/reference/summarise.html): Variablenwerte mehrerer Beobachtungen zu einem Wert reduzieren - [`group_by()`](https://dplyr.tidyverse.org/reference/group_by.html): nachfolgende Operationen werden auf Gruppen ausgeführt - `join()`: Zusammenfügen zweier Datensätze (mergen) Zu diesen Funktionen gibt es häufig Varianten und weitere Hilfsfunktionen, die z.B. bei der Variablenwahl helfen. --- # 4. Datenanalyse Datensätze die *tidy* sind, sollen für jede Beobachtung eine eigene Zeile haben und für jede Variable eine Spalte (`long`-Format). Häufig findet man aber Datensätze, die `wide` sind. Möglichkeit um von `wide` zu `long` zu kommen ist die Funktion `pivot_longer()` aus `tidyr`: ```r library(tidyr) ## version 1.0.0 data_wide <- data.frame(id = c(1,2,3,4), wage90 = c(12,13,14,11), wage95 = c(14,16,13,18)) data_long <- data_wide %>% * pivot_longer(cols = starts_with("wage"), # welche Spalten? * names_to = "year", # wie soll die neue Spalte heißen? * values_to = "wage") # welche neue Spalte enhält die Werte? class(data_long) # tbl_df, tbl, data.frame ``` --- # 4. Datenanalyse ### Eine Variable Lagemaße: ```r ## stetige Variable CPS1985 %>% summarise(mean_wage = mean(wage), sd_wage = sd(wage), med_wage = median(wage), min_wage = min(wage), q1_wage = quantile(wage,0.25)) ## für so einfache Fälle eignet sich natürlich auch: summary(CPS1985$wage) ## diskrete variable table(CPS1985$occupation) ``` --- # 4. Datenanalyse Grafisch: ```r ## lambda-Ausdruck und dot-Platzhalter CPS1985 %>% mutate(lwage = log(wage)) %>% select(lwage) %>% { lwage <- .[,1] # '.' ist der Wert von der linken Seite vor dem letzen Pipe-Operator hist(lwage) # hier also CPS1985 %>% select(wage) } ``` --- # 4. Datenanalyse ### Mehrere Variablen ```r ## mehrere stetige Variablen CPS1985 %>% select(wage, education, age) %>% { pairs(.) cor(.) } ## diskrete variablen (frequency tables) CPS1985 %>% group_by(gender,sector) %>% summarise(n = n()) ## alternativ: with(CPS1985, ftable(gender, sector) ) ## oder auch xtabs(~gender + sector, data = CPS1985) ``` --- # 4. Datenanalyse ### Aufgabe 4.1 Verwenden Sie den Datensatz `CPS1985` aus dem `AER`-Paket. 1. Fügen Sie mit `mutate()` und dem Pipe-Operator dem Datensatz die Variable `logW` (logarithmierte Löhne) hinzu. 2. Berechnen Sie nun, gruppiert nach `gender` und `occupation`, mittlere Log-Löhne sowie deren Standardabweichung. 3. Selektieren Sie jeweils für Männer und Frauen die Beobachtung mit dem höchsten Lohn. 4. Fügen Sie dem Datensatz `CPS1985` eine Variable `lowest_wage_in_occ` hinzu, welche jeweils innerhalb einer `occupation`-Gruppe konstant ist und dem niedrigsten Lohn (in der jeweiligen Gruppe) entspricht. 5. Sortieren Sie nun den Datensatz innerhalb der Geschlechtergruppen nach Löhnen. --- # 4. Datenanalyse ### Aufgabe 4.2 Erweitern Sie innerhalb des Lambda-Ausdrucks auf [Folie 8](#115) das Histogramm der (Log-)Löhne folgendermaßen: 1. Fügen Sie die Dichtefunktion einer normalverteilten Zufallsvariable, welche Mittelwert und Varianz der logarithmierten Löhne aufweist hinzu. 2. Zeichnen Sie den Mittelwert als gestrichelte Linie ein. 3. Zeichnen Sie mit einem Polygon die Fläche unter der Dichtefunktion bis zum 25%-Quantile ein. --- # 4. Datenanalyse ### Aufgabe 4.3 Erstellen Sie nun einen aktuellen Datensatz der Arbeitslosenzahlen und -Quoten in den Deutschen Kreisen (siehe [Aufgabe 2.12](#73)) indem Sie die entsprechenden Excel-Tabellen von der Hompage der [Bundesagentur für Arbeit](https://statistik.arbeitsagentur.de/Navigation/Statistik/Statistik-nach-Themen/Arbeitslose-und-gemeldetes-Stellenangebot/Arbeitslose/Arbeitslose-Nav.html) herunterladen (Arbeitslose und Arbeitslosenquoten - Deutschland, Länder, Kreise und Gemeinden). Verwenden Sie nun die Funktion `read_xlsx()` und nutzen Sie passende Filter/Selektionen um am Ende einen Datensatz zu erhalten, der drei Spalten (Region, Anzahl. Quote) und jeweils eine Zeile für jeden Landkreis/jede Kreisfreie Stadt enthält. --- # 4. Datenanalyse ### Hypothesentests Hierzu verwenden wir den Datensatz [wheat.txt](https://www.uni-regensburg.de/wirtschaftswissenschaften/vwl-tschernig/medien/programmieren-mit-r/wheat.txt), welcher Weizenpreise für die Monate März 1982 - März 2012 enhält. ```r (wheat <- read.table("wheat.txt",header=TRUE) %>% na.omit()) %>% { ## one sample t-test t.test(.$Change, alternative = "greater", mu = 0, conf.level = 0.95) ## two sample t-test t.test(Change ~ I(Year > 2000), data = ., ## t-Test-Spezifikation als 'formula' alternative = "greater", mu = 0, conf.level = 0.95) } ``` --- # 4. Datenanalyse Weitere Funktionen um Hypothesentests durchzuführen: - `var.test()`: Test auf Gleichheit der Varianzen zweier Vektoren - `chisq.test()`: `\(\chi^2\)`-Test auf Unabhängigkeit - viele weitere Pakete für spezielle Tests (`urca`, `lmtest`,...) --- # 4. Datenanalyse ### Aufgabe 4.4 - Überfliegen Sie den Aufsatz ["Incentives Work: Getting Teachers to Come to School"](http://economics.mit.edu/files/5582) von Duflo et al. Beschreiben Sie kurz das Experiment, das hier durchgeführt wurde. - Schauen Sie sich dann die [Daten](https://dataverse.harvard.edu/dataset.xhtml?persistentId=hdl:1902.1/VZJXRPURTJ) an. Die Datensätze `TreatmentSchools` und `Posttest` sind ausreichend. <br> Laden Sie die Daten in R und schauen Sie sie an. Versuchen Sie hierbei den Stata-Datensatz (Endung: .dta) mit Hilfe der `read_dta` Funktion aus dem `haven` Package zu laden. --- # 4. Datenanalyse - Erstellen Sie im Datensatz `Posttest` eine Variable `treat`, die den Wert `TRUE` annimmt für Schüler, deren School-ID (Variable `schid`) im Datensatz `TreatmentSchools` auftaucht. Verwenden Sie dazu den Operator `%in%`.<br> Beispiel: ```r a <- 1:10 a[1:10 %in% c(2,3)] ``` - Erzeugen Sie eine Teilstichprobe des Datensatzes, der die Schüler mit schriftlichem Test enthält (`post_writ==1`) - Vergleichen Sie die mittleren Testergebnisse (Variable `post_total_w`) von Treatment- und Nicht-Treatment-Schülern. - Führen Sie einen t-Test der folgenden Nullhypothese durch: "Treatment- und Nicht-Treatment-Schüler sind im Mittel gleich gut" --- # 5. Flow Control Häufig sollen Ausdrücke (Berechnungen, Schätzungen, Simulationen, Plots...) nur unter bestimmten Bedingungen und/oder sehr oft ausgeführt werden. Manchmal ist eine Operation auch so komplex, dass man sie in wiederholten Schritten leichter durchdenken und programmieren kann. Man braucht dann sog. Konstrukte: - Bedingte Ausführung (`if`, `else`) - Schleifen (`for`, `while`) - Oder, als schnellere Alternative zur Schleife die Anwendung von Funktionen auf viele Elemente gleichzeitig mit `apply()`-Funktionen verschiedener Art - `Vectorize()` modifiziert Funktionen, so dass sie auf mehrere Elemente gleichzeitig angewendet werden kann --- # 5. Flow Control ### Bedingte Ausführung ```r if ( expr ) { ## some code evaluated if is.logical(expr) == TRUE } else { ## some other code } ``` Das gesamte Konstrukt ist wieder ein Ausdruck, hat also auch einen Wert (und zwar den Wert des zuletzt von ihm ausgewerteten Ausdrucks): ```r result <- if ( 2 > 1 ) { 2 ## letzter ausgewerteter Ausdruck } else { 1 } ## result hat den wert 2 ``` --- # 5. Flow Control Vorsicht bei vektorenwertigen Ausdrücken: ```r y <- c(5, 3, 2) y > 3 if(y > 0) "Hierher!" ## wertet nur den ersten Eintrag von y aus ``` Will man prüfen, ob alle Einträge eines Vektors eine bestimmte Eigenschaft erfüllen, helfen die Quantoren `any()`/`all()`: ```r if ( all(y > 0) ) ## all() ist "TRUE", falls Argument="Vektor mit lauter TRUE" { print("Alle y sind größer als 0") } else { print("Mindestens ein Element von y ist kleiner oder gleich 0!") } ``` --- # 5. Flow Control Natürlich besteht auch die Möglichkeit, dass mehr als zwei Fälle abgedeckt werden müssen, z.B. `$$f(x) = \cases{0 \text{ für } x \leq 5\\ 4 \text{ für } 0< x \leq 5\\ 6 \text{ sonst.}}$$` ```r stepfunction <- function(x){ if ( x <= 0 ) { 0 } else if (x <= 5 ) { 4 } else { 6 } } ``` --- # 5. Flow Control Es gibt auch noch die Funktion `switch(expr, ...)`: ```r ## integer expr switch(1, "first", "second", "abc") ## -> "first" switch(2, "first", "second", "abc") ## -> "second" switch(3, "first", "second", "abc") ## -> "abc" ## character expr switch("abc", a = "first", b = "second", abc = "mix") ## -> "mix" ``` --- # 5. Flow Control Manchmal möchte man auch vektorisiert Objekte überprüfen und für jeden Eintrag einen eigenen Wert zurück erhalten, dazu gibt es die Funktion `ifelse()`: ```r x <- c(3, NA, 2) ifelse( is.na(x), "Missing", "Nicht Missing") ## absolutbetrag x <- c(-3, 5, -8, 2) ifelse( x < 0, -x, x) ``` --- # 5. Flow Control ### Aufgabe 5.1 Schreiben Sie eine Funktion `if_test` die zwei Objekte `x` und `y` entgegennimmt und überprüft ob `x` numerisch ist und `y` vom Typ character. Falls sowohl x als auch y dem entsprechen, soll die Funktione "Super" ausgeben, ansonsten mitteilen, welches der beiden Objekte `x`/`y` nicht die entsprechende Eigenschaft hat.<br> Testen Sie Ihre Funktion mit: ```r if_test(5, "char") if_test("abc", 2) if_test(list(1,2), matrix("a", 3, 3)) ``` Hinweis: nutzen Sie `is.character()` und `is.numeric()`. --- # 5. Flow Control ### Schleifen Häufig muss man kleiner Code-Bausteine öfter ausführen, hierfür existieren Schleifen. In R gibt es drei verschiedene Schleifen: - `repeat{ Code }`: Schleife, die unendlich oft `Code` ausführt, muss mit `break` beendet werden. - `while( Condition) { Code }`: der Code `Code` wird so lange ausgeführt, wie `is.logical(Condition)` dem Wert `TRUE` entspricht. - `for (value in values) { Code }`: der Code `Code` wird für jedes Element im Vektor `values` einmal ausgeführt. Dabei hat die Variable `value` beim jeweiligen Schleifendurchlauf jeweils den Wert des entsprechenden Elements Neben `break` (Abbruch der Schleife) gibt es einen weiteren Kontrollbefehl: `next` (sofortiger Sprung in den nächsten Schleifendurchlauf) --- # 5. Flow Control Beipiele ```r vec <- c("Eins","Zwei","Drei") for (v in vec) print(v) for (i in 1:10) { print(i + 2) } ``` Folgendes würde zum selben Ergebnis führen wie die letzte for-Schleife: ```r i <- 1 print(i + 2) i <- 2 print(i + 2) # ... i <- 9 print(i + 2) i <- 10 print(i + 2) ``` --- # 5. Flow Control Ausdrücke in Konstrukten werden in der jeweiligen Umgebung ausgewertet, in der sie aufgerufen werden. Damit hat die Variable `i` nach dem Durchlaufen der Schleife ```r for (i in 1:100) { ## some code using the variable i } ``` den Wert 100. Alle diese Konstrukte können natürlich beliebig kombiniert werden: ```r control <- TRUE while (control){ if (a > b){ for (i in seq(along = longObject)){ ## do something with i } if (error) break } else { next } } ``` --- # 5. Flow Control ### Beispiel: numerische Optimierung einer Funktion Algorithmische Idee (sehr naiv, daher nicht für die Praxis geeignet):<br> Wir wollen numerisch das Maximum einer nach unten geöffneten Parabel finden. Wir starten bei einem Wert, wo wir sicher wissen, dass die Parabel noch ansteigt, bewegen uns schrittweise nach rechts überprüfen dabei, ob der Wert der Parabel noch ausreichend größer wird. Ist dies nicht der Fall, stoppen wir: .center[ 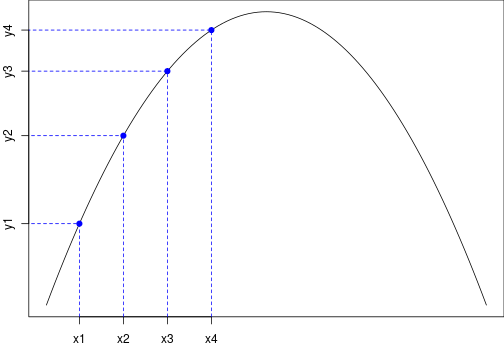<!-- --> ] --- # 5. Flow Control Code: ```r parabel <- function(x) -x^2 xx <- -2 ## hier starten wir yy <- parabel(xx) ## welchen Wert hat die Parabel hier? dY <- 1 ## wir initialisieren einen Wert, damit while() nicht gleich abbricht while( dY > 0.0000001){ xx <- xx + 0.01 ## neuen xx einen schritt weiter rechts yyNew <- parabel(xx) ## welchen wert hat die Parabel an dem neuen xx? dY <- yyNew - yy ## um wieviel ist der Wert der Parabel durch diesen Schritt ## angestiegen? dieses dY wird vor dem nächsten ## Schleifendurchlauf benutzt um zu überprüfen ob die ## Bedingung dY >0.0000001 noch erfüllt ist. yy <- yyNew ## mach das yy für den nächsten Schleifendurchlauf zum neuen Wert } ``` --- # 5. Flow Control ### Aufgabe 5.2 1. Berechnen Sie das Matrixprodukt aus A und B ```r A <- matrix(1:12,4,3) B <- matrix(1:9,3,3) C <- A %*% B ``` ohne den Operator `%*%`. Verwenden Sie eine oder mehrere Schleifen und etwa <br>`sum(A[1,] * B[,1])` für `C[1,1]`, usw. 2. Erstellen Sie nun aus den Schleifen der vorigen Teilaufgabe eine Funktion `mat_mult`, die bei Eingabe zweier Matrizen das Produkt berechnet, aber vorher mit if testet, ob die Zeilenlänge der einen Matrix die Spaltenlänge der anderen Matrix ist. Testen Sie das mit den Matrizen von oben. --- # 5. Flow Control ### Aufgabe 5.3 - Schreiben Sie eine Funktione `optimizer`, welche bei Übergabe einer (rellwertigen) Funktion numerisch nach dessen Optimum sucht. Nutzen Sie dazu den obigen Beispielcode. - Versuchen Sie nun anstelle des obigen Optimierungsverfahrens ein Bisektionsverfahren zu implementieren. Sehen Sie sich dazu den Wikipediaartikel zum [Bisektionsverfahren](https://de.wikipedia.org/wiki/Bisektion) an.<br> Hinweis: Um die Ableitung einer Funktion `fun` an der Stelle `x` zu berechnen, können Sie die Funktion `numeriDeriv()` aus dem Paket `stats` folgendermaßen nuzten: ```r env.o2 <- new.env() dfun <- function(x) { assign("x",x, envir = env.o2) attr(numericDeriv(quote(fun(x)), "x", rho = env.o2), which = "gradient")[1,1] } ``` --- # 5. Flow Control ### apply- Konstrukte Schleifen in R sind relativ langsam führen auch manchmal zu nicht sehr gut lesbarem Code. Wo es möglich ist, sollten Schleifen vermieden werden. Häufig lässt sich eine Schleife durch Vektorisierung ersetzen (mit extremen Performance-Gewinnen, dann werden nämlich Schleifen in C und nicht in R aufgerufen) In R gibt es darüber hinaus eine Familie an `apply`-Konstrukten, welche gewissermaßen vektorisiertes Arbeiten ermöglichen. --- # 5. Flow Control Zur Arbeit mit Matrizen und Arrays gibt es die Funktion `apply()`. Mit dieser Funktion können auf Spalten oder Zeilen geeignete Funktionen angewandt werden. ```r A <- matrix(c(2, 6, 3, 4, 5, 7, 8, 4, 1), ncol = 3) ## zeilen-Maximum: apply(X = A, ## ein Array oder eine Matrix MARGIN = 1, ## Über welche Indizes soll die Funktion angewandt werden ## (1-> Zeilen, 2-> Spaltem) FUN = max ## welche Funktion soll angewandt werden? ) X <- rnorm(1000) %>% matrix(ncol = 10) apply(X, 2, var) ## Varianzen der Spalten ``` --- # 5. Flow Control Häufig liegen Listen vor, und man möchte jeweils auf die Elemente der Liste eine Funktion anwenden und die Ergebnisse wieder in einer Liste zusammenfassen. Dafür gibt es die Funktion `lapply()` ```r Liste <- list(a=c(4,8,7), b=seq(0,100,5), c=c(TRUE,TRUE,FALSE,TRUE) ) ListeSumme <- lapply(Liste, sum) class(ListeSumme) ## wieder eine Liste ``` Häufig braucht man nicht als Ergebnis eine Listenstruktur, sondern beispielsweise die Ergebnisse in einem Vektor (oder einer Matrix/einem Array) zusammengefasst. Dies mach `sapply()` ```r ListenSumme <- sapply(Liste, sum) class(ListenSumme) ## "numeric" ``` --- # 5. Flow Control Alle Mitglieder der apply-Familie akzeptieren das `...`-Argument. Hier können Parameter spezifiziert werden, die an die übergebene Funktion weitergereicht werden: ```r ## eigene Funktion funnyFun <- function(x, m, std) sum(x)/( 2 * rnorm(1, mean=m, sd=std)) ## über ... werden 'm' und 'std' an funnyFun weitergereicht: sapply(X=Liste, FUN=funnyFun, m=2, std=5) ## auch Plots lassen sich so erstellen: par(mfrow=c(length(Liste),1)) lapply(Liste, plot, main="Titel", type="l", lwd=2) ``` --- # 5. Flow Control ### Aufgabe 5.4 Erstellen Sie eine `\(10\times 10\)` Matrix, in der als `\(ij\)`-tes Element `\((i\cdot j)^{(1/j)}\)` steht. Benutzen Sie hierfür zwei ineinandergeschriebene Schleifen. Berechnen Sie dann die Spalten- und Zeilensummen dieser Matrix mit einer apply-Funktion Ihrer Wahl. --- # 5. Flow Control ### Weitere hilfreiche Sprachelemente Manchmal möchte man dynamisch benannte (abhängig von anderen Variablen) Objekte erzeugen und später wieder deren Wert bestimmen. Dies geht mit `<-` nicht. Es gibt deshalb die Funktionen `assign()` und `get()`. ```r objectName <- "x" ## Die folgenden Zuweisungen sind äquivalent: x <- 2 assign("x", 2) assign(objectName, 2) ## Den Wert des Objektes "x" ausgeben get(objectName) ## -> 2 ``` --- # 5. Flow Control Weiteres Beispiel ```r ## je mach Wert der Variable x soll entweder mean() oder sum() ausgeführt werden myFunc1 <- mean myFunc2 <- sum x <- 1 get(paste("myFunc",x,sep=""))(1:10) ## mean() x <- 2 get(paste("myFunc",x,sep=""))(1:10) ## sum() ``` --- # 5. Flow Control Weiteres Beispiel für Nützlichkeit von `assign()`: <br> speichere den `summary()`-Output von jeder Variable des Datensatzes `CPS1985` in `summary_{Variablenname}` ```r names(CPS1985) for(variable in names(CPS1985)) { assign(paste("summary_", variable, sep = ""), summary(get(variable))) print(paste("summary_", variable, sep = "")) } ``` Was genau passiert hier? --- # 5. Flow Control Zerlege in einzelne Schritte: ```r ## Schleife läuft über die Werte: names(CPS1985) ## Erster Durchlauf: variable <- names(CPS1985)[1] variable get(variable) summary(get(variable)) paste("summary_", variable, sep = "") assign(paste("summary_", variable, sep = ""), summary(get(variable))) ``` --- # 5. Flow Control ### Fehlerbehandlung Häufig weiß man im Voraus nicht, ob die Ausführung von Code zu Fehlern führt. - eine Möglichkeit: schon vor der Ausführung alle Fälle zu prüfen, die zu Fehlern führen können - eine weitere (pragmatische) ist die Verwendung von in vielen Programmiersprachen vorhandenen Sprachelementen zur **Fehlerbehandlung**: ```r value <- tryCatch({ ## some code to be evaluated expr }, warning = function(w){ ## code evaluated if evaluating of expr leads to a warning print(w) }, error = function(e){ ## code evaluated if evaluating of expr leads to an error print(w) }) ``` --- # 5. Flow Control ### Rekursion Manchmal ganz hilfreich: rekursive Funktionen ```r faculty <- function(n){ if (n<0) stop("Faculty only for positive integers") if (n<2) return(1) n * faculty(n-1) } ``` --- # 5. Flow Control ### Aufgabe 5.5 1. Ersetzen Sie die Schleife über variable mit einem apply-Befehl 2. Wie lässt sich für das vorliegende Problem die Verwendung von assign und get vermeiden? ### Aufgabe 5.6 Schreiben Sie eine Funktion mit Namen "fib", die bei Eingabe einer natürlichen Zahl `n` die `n`-te Fibonaccizahl berechnet. Die Fibonaccizahlen sind eine rekursive Folge mit `\(a_0=0\)`, `\(a_1=1\)` und `\(a_n=a_{n-1}+a_{n-2}\)`. --- # 5. Flow Control ### Aufgabe 5.7* Untersuchen Sie grafisch die Hypothese der Einkommenskonvergenz zwischen den deutschen Kreisen und kreisfreien Städten. Besorgen Sie von [https://www.regionalstatistik.de](https://www.regionalstatistik.de) das verfügbare Einkommen 2000 und möglichst aktuell. Sie können die Tabellen als Text kopieren, in eine Datei speichern und in R importieren. Erstellen Sie aussagekräftige Grafiken, etwa die Veränderung des Einkommens auf der y-Achse vs. das Anfangsniveau auf der x-Achse. Führen Sie die Untersuchung auch auf Teilpopulationen durch. --- # 6. Regression In der Ökonometrie (natürlich übertragbar auf andere Wissenschaftsgebiete) fragt man sich häufig - Findet Einkommenskonvergenz statt (führt geringes Einkommensniveau zu c.p. höherem Wachstum in der Zukunft?) - Ist Bildung eine wichtige Determinante des Wachstums? - Beschleunigt Außenhandel das Wachstum? - Ist politische Stabilität wichtig / behindert Gewalt den ökonomischen Wohlstand? - Ist ceteris paribus in Afrika das Wachstum langsamer? Klassisches Instrumentarium dafür: **multiples lineares Regressionsmodell** `$$y_i = \beta_0 + \beta_1 x_{1,i} + \beta_2 x_{2,i} + \dots \beta_p x_{p,i} + u_i,\ i=1,\dots,n$$` bzw. in Matrixschreibweise: `$$\mathbf{y} = \mathbf{X}\beta + \mathbf{u},$$` wobei `\(\mathbf{y}, \mathbf{u}\in\mathbb{R}^{n}\)`, `\(\mathbf{X}\in\mathbb{R}^{n\times k}\)`, `\(\beta\in\mathbb{R}^{k}\)` und `\(k = p + 1\)`. --- # 6. Regression ### Formel-Objekte R wurde entworfen, um relativ einfach verschiedene statistische Modelle schätzen zu können. Es existiert daher in R eine eigene Objektklasse um statistiche Modelle symbolisch zu beschreiben, sogenannte `formula`-Objekte. Siehe dazu `?formula`. Beispiel: das Regressionsmodell `\(y_i = \beta_0 + \beta_1 x_{1,i} + \beta_2 x_{2,i} + u_i\)` lässt sich als `formula` folgendermaßen angeben: `y ~ x1 + x2` --- # 6. Regression - Wichtige Operatoren: - `~`: Basis für alle Modelle `y ~ model` gibt an, dass die abhängige variable `y` durch die linearen Prädiktoren beschrieben in `model` modelliert wird. - `+`: Modelle bestehen aus durch `+` getrennte Terme (im Einfachen Fall sind das Variablen) - `:`: Erzeugt Interaktionsterme der Variablen - `*`: `a*b` ist gleichbedeutend zu `a + b + a:b` - `^`: `(a + b)^2` ist äquivalent zu `(a + b)* (a + b)` - Achtung: `y ~ x1 + x2^2` ist nicht das Modell `\(y_i = \beta_0 + \beta_1 x_{1,i} + \beta_2 x^2_{2,i} + u_i\)`! - Für arithmetische ausdrücke (z.B. Transformationen von Variablen) gibt es die Funktion `I()`: <br>`y ~ x1 + I(x2^2)` - Faktorvariablen in formeln werden automatisch *dummy-codiert* --- # 6. Regression Standardfunktion um lineare Modelle zu schätzen ist `lm()`. Diese Funktion gibt ein `lm`-Objekt zurück mit einer Vielzahl an Methoden: `summary()`, `plot()`, `predict()`,... ```r library(AER) data(GrowthSW) growth.eq1 <- lm(growth ~ rgdp60, data=GrowthSW) summary(growth.eq1) ``` --- # 6. Regression Output ``` ## ## Call: ## lm(formula = growth ~ rgdp60, data = GrowthSW) ## ## Residuals: ## Min 1Q Median 3Q Max ## -4.6309 -1.0485 0.0831 0.8722 5.3183 ## ## Coefficients: ## Estimate Std. Error t value Pr(>|t|) ## (Intercept) 1.796e+00 3.780e-01 4.751 1.21e-05 *** ## rgdp60 4.735e-05 9.494e-05 0.499 0.62 ## --- ## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1 ## ## Residual standard error: 1.908 on 63 degrees of freedom ## Multiple R-squared: 0.003932, Adjusted R-squared: -0.01188 ## F-statistic: 0.2487 on 1 and 63 DF, p-value: 0.6197 ``` --- # 6. Regression Mehrere Variablen, Transformation: ```r growth.eq2 <- lm(growth ~ rgdp60 + tradeshare + * education + I(education^2) + # education linear und im quadrat revolutions + assassinations, data = GrowthSW) sum.eq2 <- summary(growth.eq2) ``` Ohne Konstante: ```r growth.eq3 <- lm(growth ~ -1 + rgdp60 + tradeshare, data = GrowthSW) ``` Alle Variablen im Datensatz: ```r growth.eq4 <- lm(growth ~ ., data = GrowthSW) ``` Mit `-` lassen sich auch Variablen wieder entfernen: ```r growth.eq5 <- lm(growth ~ . -rgdp60, data = GrowthSW) ``` --- # 6. Regression Mit Faktorvariable: ```r GrowthSW <- GrowthSW %>% tibble::rownames_to_column("country") %>% mutate(continent = factor(ifelse( country %in% c("Zaire","Niger","Senegal", "Zimbabwe" ,"Togo", "South Africa", "Sierra Leone" ,"Ghana","Kenya", "Mauritius"), "africa", "other"), levels = c("africa", "other"))) growth.eq6 <- lm(growth ~ rgdp60 + tradeshare + continent, data = GrowthSW) summary(growth.eq6) ``` Interaktionen: ```r growth.eq7 <- lm(growth~ * log(rgdp60) * continent + tradeshare+log(education)+ revolutions+assassinations, data=GrowthSW) sum.eq7 <- summary(growth.eq7) ``` --- # 6. Regression ### Zugriff auf Regressionsergebnisse - entweder mit den Zugriffsfunktionen `coefficients()`, `fitted()`, `residuals()`,... - oder Zugriff auf das Listenobjekt `lm` oder `summary.lm`: ```r str(growth.eq7) ## structure of lm object str(sum.eq7) ## structure of summary.lm object # elements in lm object growth.eq7$coefficients growth.eq7$residuals growth.eq7$fitted # summary.lm offers more stuff: sum.eq7$coefficients ## matrix with estimate, std-dev, t-stat, pvalue in colums sum.eq7$sigma ## estimate of residual standard error sum.eq7$adj.r.squared ## R^2 of regression ``` --- # 6. Regression ### Modellwahl Zur beantortung der Frage, welches Modell "besser" ist werden häufig Modellselektionskriterien verwendet (AIC, BIC, ...)? Vorgehen: Modell mit dem kleinsten Wert ist "bestes" Modell. ```r # Akaike Kriterium AIC(growth.eq5) # kleiner = besser AIC(growth.eq1) # Schwarz Kriterium BIC(growth.eq5) # besser BIC(growth.eq1) ``` --- # 6. Regression Im Paket `MASS` gibt es die Funktion `stepAIC`, mit der schrittweise, ausgehend von einem großen Modell, ein "optimales" Modell gefunden werden kann: ```r install.packages("MASS") library(MASS) ?stepAIC growth.eqAIC <- stepAIC( lm(growth ~ (log(rgdp60) + tradeshare + log(education)+ revolutions + assassinations + continent)^2, data=GrowthSW)) ``` --- # 6. Regression ### Aufgabe 6.1 Nutzen Sie den Growth Datensatz von oben. 1. Testen Sie ein paar Möglichkeiten für die Formeleingabe: - ohne/mit Konstante - Quadratisch, Kubisch, den doppelten Regressor - Interaktionen (von Gruppen) - stepAIC Befehl benutzen 2. Nehmen Sie nun eines Ihrer Modelle und wenden Sie die summary()-Methode darauf an. Sprechen Sie Schätzungen, Standardfehler, p-values, die Residuenquadratsumme und den angepassten `\(R^2\)` Wert an. --- # 6. Regression ## Modelldiagnose und Tests ### 1. Heteroskedastie Graphisch: ```r uhat_AIC <- resid(growth.eqAIC) # growth.eqAIC$residuals yhat_AIC <- fitted(growth.eqAIC) plot(yhat_AIC,uhat_AIC^2, main="Squared Residuals vs. Fitted Values") ``` 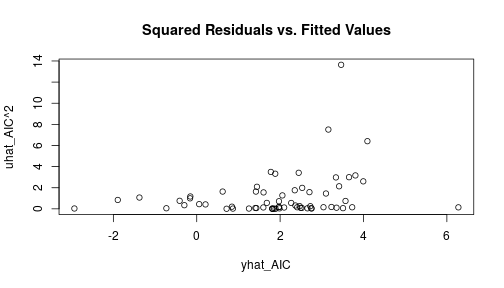<!-- --> --- # 6. Regression Test auf Heteroskedastie (Breusch-Pagan, White): ```r # Breusch - Pagan bptest(growth.eqAIC) # White Test summary(lm(I(uhat_AIC^2)~ yhat_AIC + I(yhat_AIC^2)) )$r.squared * length(yhat_AIC) %>% pchisq(df = 2, lower.tail = FALSE) ``` Ausweg: Heteroskedastie-robuste Varianz-Kovarianzmatrix ```r vcov.robust <- vcovHC(growth.eqAIC, type = "HC") coeftest(growth.eqAIC,vcov=vcov.robust) ``` --- # 6. Regression ### Modelldiagnose: 2. Nichtlinearität ```r plot(yhat_AIC,uhat_AIC,main="Residuals vs. Fitted Values") ``` 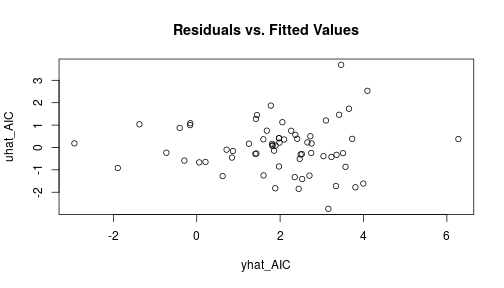<!-- --> --- # 6. Regression RESET-Test: ```r resettest(growth.eqAIC) ``` Weitere Diagnoseplots: ```r plot(growth.eqAIC) ``` --- # 6. Regression ### Hypothesentests - Testentscheidungen zu einfachen Hypotesen der Form `\(H_0: \beta_j = 0\)` lassen sich direkt am Output ablesen - genauso: Overall-F-Test - weitere Hypothesen (z.B `\(H_0: \beta_j = 3\)`) oder zusammengesetzte Hypothesen lassen sich eher mit der Funktion `linearHypothesis()` aus dem Pakte `car` überprüfen: ```r ## symbolisch: linearHypothesis(growth.eqAIC, "log(education)=2") ## Andere Form der Hypothese: R %*% beta = r (hier ist R ein 1xk-Vektor, r skalar) R <- c(0,0,0,1,0,0,0,0,0) # 4. Eintrag wählt 4. Koeffizienten r <- 2 # .. und der soll 2 sein laut H_0 linearHypothesis( growth.eqAIC , hypothesis.matrix =R, rhs=r ) ## Heteroskedastierobuster Test: linearHypothesis(growth.eqAIC,"log(education)=2",vcov=vcov.robust) ``` --- # 6. Regression ### Tests von mehreren Hypothesen (F-Tests) ```r ## symbolisch linearHypothesis(growth.eqAIC, c("log(rgdp60)=0","log(rgdp60):assassinations=0")) ## Mit der Formulierung R %*% beta = r: R <- matrix(c(0,1,rep(0,7), rep(0,7),1,0),nrow=2, byrow=TRUE) r <- c(0,0) linearHypothesis(growth.eqAIC,R,r) ``` ### Konfidenzintervalle ```r confint(growth.eqAIC,parm=c("revolutions"),level=0.9) ``` --- # 6. Regression ## Prognose ```r ## Neue Beobachtungen X0 <- data.frame( growth=c(NA,NA), revolutions=c(0.3,0.13), rgdp60=c(2000,6000), tradeshare=c(0.3,0.7), continent=factor(c("africa","other"), levels=c("other","africa")), assassinations=c(0.4,0.2), education=c(2,4) ) rownames(X0) <- c("Land 1", "Land 2") ## Punktprognose, Prognoseintervall, etc. predict(growth.eqAIC, newdata=X0, se.fit=TRUE, interval="prediction") ``` --- # 6. Regression ### Aufgabe 6.2 - Benutzen Sie die Daten `CPS1985` um ein ad-hoc Regressionsmodell für den logarithmierten Stundenlohn zu spezifizieren und zu schätzen. - Führen Sie nun ausgehend von einem sehr allgemeinen Modell eine Modellwahl durch, etwa durch jew. Entfernen des am wenigsten signifikanten Regressors. Testen Sie danach die dabei verwendeten Ausschlussrestriktionen.<br> Hinweise: - Allgemeines Modell: Formel in Quadrat für alle möglichen Interaktionen - Schleife mit Abfrage, ob der maximale p-value größer als 5% ist. - Löschen dieses Regressors aus dem Modell. Wichtig: Ausgabe der Matrix X im Modell mit `lm(y~x, x=TRUE)`, da dann das Löschen des jeweiligen Regressors einfacher ist. Die Designmatrix `X` kann dann über `a$x`, wobei `a <- lm(y~x, x=TRUE)`, aufgerufen werden. - Alternativ: Einfügen einer neuen Formel, Problem aber bei Interaktionen. --- # 6. Regression - Versuchen Sie nun die beiden Modelle anhand geeigneter Diagnosetests, Kriterien Grafiken etc. zu vergleichen. Entscheiden Sie sich für ein Modell. - Welchen Lohn hätte derjenige Angestellte zu erwarten, der bezüglich aller erklärenden Charakteristika dem Median entspricht. Welchen der "Mittelwertarbeitnehmer"?<br> Hinweis: nehmen Sie hierfür ein Modell, das lediglich numerische erklärende Variablen enthält! - Geben Sie jeweils ein Prognoseintervall an. --- # 6. Regression ### FGLS bei Heteroskedastie (gewichtete Regression) ```r # Erzeuge (inverse) Gewichte (geschätzte Varianz des Fehlers einer Beobachtung) var.eq <- lm(log(uhat_AIC^2)~.-growth,data=GrowthSW) summary(var.eq) sigma_sq_hat <- exp(fitted(var.eq)) # Gewichte mit "weights"-Argument verwenden growth.eqFGLS <- update(growth.eqAIC,weights=1/sigma_sq_hat) summary(growth.eqAIC) summary(growth.eqFGLS) ``` --- # 6. Regression ### Dynamische Regression (Beispiel Philippskurve) Speichere dazu den Datensatz [dataUS](https://www.uni-regensburg.de/wirtschaftswissenschaften/vwl-tschernig/medien/programmieren-mit-r/dataus.csv) lokal unter `data_us.csv` ab. ```r data.us <- read.csv("data_us.csv")[,-1] PhilData <-ts( subset(data.us,select=c("inf","ur")), start=c(1948,1), frequency=4) philCurve <- dynlm( inf ~ ur + L(ur,3) + L(inf,1:3) ,data=PhilData) summary(philCurve) ``` --- # 7 Monte Carlo - Verfahren, um analytisch schwierig zu lösende Probleme mit Hilfe von Zufallsexperimenten (also Wahrscheinlichkeitstheorie) numerisch (beliebig genau) zu approximieren - Grundlage ist das Gesetz der Großen Zahlen:<br> Seien `\(X_i\sim iid(\mu, \sigma^2), i = 1,\dots,n\)` mit `\(\mu<\infty\)`, dann gilt: $$ \underset{n\to\infty}{\operatorname{plim}}\frac{1}{n}\sum_{i=1}^nX_i = \mu$$ --- # 7 Monte Carlo ### Beispiel: Berechnung der Kreiszahl `\(\pi\)` Erinnerung: `\(r^2 \pi\)` ist die Fläche eines Kreises, also `\(\frac{1}{4}\pi\)` die Fläche des Viertelkreises in einem Einheitsquadrat.<br> Erzeugt man nun eine Anzahl `\(n\)` von gleichverteilten Punkten in diesem Quadrat, so kann man mit Vergrößerung dieser Anzahl `\(\pi\)` immer näher als den vierfachen Anteilswert der Punkte im Viertelkreis bestimmen. ### Vorgehensweise: Ziehe aus der bivariaten Gleichverteilung `\(n\)` Punkte im Einheitsquadrat. Zähle anschließend die Anzahl `\(m\)` der Punkte, deren Abstand zum Ursprung kleiner als eins ist (also Punkte mit der Eigenschaft `\((x^2+ y^2 < 1)\)`, wobei `\(x\)` und `\(y\)` jeweils die Koordinaten der Punkte sind. `\(4\frac{m}{n}\)` ist dann eine Monte-Carlo-Näherung für `\(\pi\)`. --- # 7 Monte Carlo ```r # Zufallsgenerator setzen: set.seed(1234) # Anzahl der gleichverteilten Punkte n <- 1000 # Benötigen Punktepaare (x,y) im Einheitsquadrat mit Länge = Breite = 1 x <- runif(n, min=0, max=1) y <- runif(n, min=0, max=1) plot(x,y) # Einzeichnen des Viertelkreises: # Aus x^2+y^2=1 folgt f(x)=sqrt(1-x^2) z <- seq(0,1,0.01) lines(z, sqrt(1-z^2), col="red") # Berechnung des Anteils der Punktepaare, bei denen x^2+y^2 <=1 counter <- x^2+y^2<=1 inCircle <- sum(counter) portion <- inCircle / n pi <- 4* portion pi # wahres pi: 3.14159265359... ``` --- # 7 Monte Carlo Etwas besser wäre natürlich eine Funktion, die in Abhängigkeit von `\(n\)` die Approximation von `\(\pi\)` ausgibt: ```r PiApproxMC <- function(n) { x <- runif(n, min=0, max=1) y <- runif(n, min=0, max=1) # Berechnung des Anteils der Punktepaare, bei denen x^2+y^2 <=1 portion <- mean(x^2+y^2<=1) pi <- 4*portion/n return(pi) } piEst <- PiApproxMC(2000) ``` --- # 7 Monte Carlo Wie hängt der Wert pi von Anzahl der verteilten Punkte ab? ```r n <- matrix(seq(10000,100000, 10000), ncol=1) points <- apply(n, MARGIN=1, FUN=PiApproxMC) plot(n, points) abline(h=3.14159265359) mean(points) -3.14159265359 ``` --- # 7 Monte Carlo ### Weiteres Beispiel: Simulation zur Evaluation von Schätzeigenschaften bei Modellselektion Beispiel aus [Leeb und Pötscher (2005) "Model Selection: Facts and Fiction"](http://journals.cambridge.org/action/displayFulltext?type=1&fid=280990&jid=ECT&volumeId=21&issueId=01&aid=280988) Frage:<br> Welchen Einfluss hat Modellwahl auf die Schätzeigenschaften im Regressionsmodell mit 2 Regressoren? DGP: `\(y = \alpha\,x_1 + \beta\, x_2 + u\)` Man sei nur an `\(\alpha\)` interessiert und muss entscheiden, ob `\(x_2\)` ins Modell gehört oder nicht!<br> Zielkonflikt:<br> `\(x_2\)` weglassen `\(\Rightarrow\)` kleine Schätzvarianz für `\(\alpha\)`, aber evtl. omitted variable bias! Idee: Schätzen von beiden Modellen für viele Beobachtungen mit festen Regressoren, und simulierten Fehlern --- # 7 Monte Carlo Seed und Parameter festlegen und Ziehen der Regressoren `\(X\)`: ```r set.seed(333) alpha <- 1 beta <- .5 reps <- 1000 # Anzahl Replikationen n <- 80 # Stichprobengröße rho <- 0.8 # Korrelation zwischen x1 und x2 sig <- c(sdx1=1,sdx2=1,sdu=1) # Stds von x1, x2 und u # Berechne Kovarianzmatrix von (x1, x2) = 2x2 Matrix mit den vier Einträgen CovX <- matrix(c(sig[1]^2, sig[1]*sig[2]*rho, sig[1]*sig[2]*rho, sig[2]^2), ncol = 2) # EINE (!) n x 2 Matrix X wird ereugt, die sofort mit der Wurzel der Korrelationsstruktur, # also der Choleski Zerlegung der Varianz-Kovarianzmatrix multipliziert wird (X <- matrix(rnorm(2*n),n) %*% t(chol(CovX))) ``` ``` ## [,1] [,2] ## [1,] 0.252425039 0.25142751 ## [2,] 2.670761982 0.55206074 ## [3,] -3.009689050 -0.71879944 ## [4,] -0.867111535 -0.85863788 ## [5,] -1.861076144 -0.25133666 ## [6,] -0.082291562 0.14015404 ## [7,] 1.356138802 0.09231369 ## [8,] 1.052694315 0.31431493 ## [9,] 0.425516917 0.05459959 ## [10,] -1.628013163 -0.80064801 ## [11,] -0.365711123 0.56939187 ## [12,] -0.643359460 0.17320812 ## [13,] 0.800401892 0.56740520 ## [14,] 0.599718288 0.88777007 ## [15,] -1.151286097 -0.24599064 ## [16,] 0.081113976 -0.02210912 ## [17,] 0.645741090 0.21075998 ## [18,] -1.401384994 -1.09131450 ## [19,] 1.344579876 -0.01806935 ## [20,] -0.072491850 0.23097404 ## [21,] -0.831207634 -0.09366545 ## [22,] 1.379330527 0.30934047 ## [23,] -1.232573939 -0.39180559 ## [24,] 1.649586132 1.28156984 ## [25,] -0.302591183 -0.05373583 ## [26,] -0.339886925 -0.77391553 ## [27,] 0.347176536 -0.75723477 ## [28,] 1.562902426 -0.16936531 ## [29,] -0.014159795 -0.31865671 ## [30,] -2.060298797 0.09566693 ## [31,] -0.723858115 -0.01580149 ## [32,] 0.092841199 -0.15526722 ## [33,] 1.061090813 -0.06628415 ## [34,] -0.602410253 -0.22557657 ## [35,] 1.130862596 1.25865146 ## [36,] 0.002179392 0.29068765 ## [37,] 0.407924881 0.09866464 ## [38,] 0.363910311 0.15903712 ## [39,] 0.084748043 0.38709662 ## [40,] -1.486443202 0.28804264 ## [41,] 0.199627609 0.09247076 ## [42,] 0.145237873 0.28169537 ## [43,] -0.668953185 -0.73981352 ## [44,] -2.447498082 -0.64111026 ## [45,] 1.388402572 1.17775740 ## [46,] 2.939603882 0.88759661 ## [47,] 0.077033924 -0.02768655 ## [48,] 0.333129566 0.46897099 ## [49,] 2.139964708 0.28940366 ## [50,] 2.035887871 0.16972749 ## [51,] -0.692887336 -0.39436384 ## [52,] -0.233882227 -1.10637615 ## [53,] -0.131029837 -0.31585052 ## [54,] 0.066610513 0.09025054 ## [55,] -1.888542635 -1.47213556 ## [56,] 0.475131446 0.44214401 ## [57,] -1.234968152 0.14913610 ## [58,] 0.191583525 0.40024383 ## [59,] 0.967839097 0.39925088 ## [60,] 2.265902502 1.47969717 ## [61,] -0.791989437 0.05087134 ## [62,] 1.764689649 0.53084724 ## [63,] 1.209296860 -0.10498796 ## [64,] 1.434859323 0.96608890 ## [65,] 1.476278728 0.49836645 ## [66,] -0.632811374 0.66808631 ## [67,] -0.597449020 1.04582237 ## [68,] 1.873002570 0.29426161 ## [69,] -1.300867716 0.27414227 ## [70,] -1.239242403 -0.08024850 ## [71,] 0.144763553 0.68120707 ## [72,] -0.330236826 0.15968118 ## [73,] -0.317694356 0.37790954 ## [74,] 0.751487551 0.38833233 ## [75,] -1.290035544 -0.41671493 ## [76,] -2.048020750 -1.25249049 ## [77,] -0.546653980 0.10755813 ## [78,] 2.611114485 0.72308468 ## [79,] 0.811442527 -0.03602403 ## [80,] 0.837337765 0.35136214 ``` --- # 7 Monte Carlo Initialisieren von Leerobjekten in welche später die geschätzten Parameter abgespeichert werden ```r # Initialisierung der Vektoren für die geschätzten alphas alpha_u <- numeric(reps) # unrestringiertes Modell mit zwei Regressoren alpha_r <- numeric(reps) # restringiertes Modell mit einem Regressor alpha_sc <- numeric(reps) # Vorher mit Schwarz Kriterium gewähltes Modell ``` In der eigentlichen Simulation wird der Prozess nun `reps` mal wiederholt, wobei die variable `u` und somit auch variable `y` jedes mal neu zu Beginn eines Schleifendurchlaufs erzeugt werden --- # 7 Monte Carlo ```r for (i in 1:reps){ # Simulation von u, y und Erstellen eines Datensatzes (alpha=1, beta=1 !) u <- rnorm(n,sd=sig[3]) data <- data.frame(y=alpha*X[,1]+beta*X[,2]+u,x1=X[,1],x2=X[,2]) # Schätzen des unrestringierten und restringierten Modells U <- lm(y~ -1 + x1 + x2, data = data) R <- lm(y~ -1 + x1, data = data) # Koeffizientenschätzer für alpha im jeweiligen Modell alpha_u[i] <- U$coef[1] alpha_r[i] <- R$coef[1] # Falls der BIC Wert des unrestringierten Modells kleiner ist, # als der des restringierten Modells, soll die unrestringierte #Sschätzung in alpha_sc gespeichert werden, ansonsten die andere alpha_sc[i] <- if (AIC(U,k=log(n)) < AIC(R,k=log(n))) U$coef[1] else R$coef[1] } ``` --- # 7 Monte Carlo Graphische Auswertung der Simulation: ```r # Plotten der Dichte der Schätzkoeffizienten plot(density(alpha_r , adjust=1.2), xlim = range(c(alpha_u,alpha_r, alpha_sc)), main = "Density of OLS estimator: Model Selection Effects", col = "blue") lines(density(alpha_u, adjust=1.2), col = "darkgreen") lines(density(alpha_sc, adjust=1.2), col = "red") text(x = alpha+.02, y = 0.2, expression(alpha[0])) text(x = alpha+.02, y = 2, expression(alpha[sc]), col = "red") text(x = alpha+.02, y = 3.1, expression(alpha[u]), col = "darkgreen") text(x = alpha, y = 1, expression(alpha[r]), col = "blue") abline(v = alpha, lty = 4) legend("topleft", inset = 0.01, legend = c("Restricted Model","Unrestricted Model","Model Selection"), lty = c(1,1,1), col = c("blue","darkgreen","red")) ``` --- # 7 Monte Carlo <!-- --> --- # 7 Monte Carlo Weitere Auswertungen ```r # Verzerrung: Omitted Variable Bias im restringierten Modell UND im SC gewählten Modell mean(alpha_r) mean(alpha_u) mean(alpha_sc) # Varianz: Restringiertes Modell am besten var(alpha_r) var(alpha_u) var(alpha_sc) # MSE: MSE beim unrestringierten Modell am niedrigsten mean((alpha_r-1)^2) mean((alpha_u-1)^2) mean((alpha_sc-1)^2) ``` --- # 7 Monte Carlo ### Simulationsbasierte Tests (Monte Carlo Test) Anwendung, wenn unter `\(H_0\)` die Verteilung der Teststatistik simuliert werden kann (pivote Teststatistik, oder bei asymptotisch pivoten Tests: Dickey/Fuller etc.). Dann können die kritischen Werte, p-values etc. simuliert werden auch wenn sie nicht in einer Tabelle stehen.<br> Fazit: Oft weiß man Verteilungen nicht, dann gibt es aber immer noch die Möglichkeit mittels Simulation die Verteilungen der Teststatistiken zu simulieren Beispiel `\(\chi^2\)`-Anpassungstest (Statistik II - siehe Formelsammlung, Wikipedia "Chi-Quadrat-Test" -> "Verteilungstest")<br> Frage: War die Notenverteilung der Klausur gleichverteilt? ```r n <- 37 # Teilnehmer k <- 13 # Notenklassen x <- c(1,7,3,3,2,3,3,5,2,4,2,2 # Beobachtete Notenverteilung x0 <- rep(37/13,13) # Theoretische Notenverteilung bei Gleichverteilung test_stat <- sum((x-x0)^2/x0) # Teststatistik des Xi^2 Anpassungstests pchisq(test_stat,df=k,lower.tail=FALSE) # P-Value (asymptotisch) ``` --- # 7 Monte Carlo Aber: `\(\chi^2\)`-Verteilung gilt nur asymptotisch, die Verteilung unter `\(H_0\)` lässt sich aber exakt bestimmen: per Simulation! Aufgabe nun: Simulieren von 9999 Realisationen der Teststatistik bei Gültigkeit von `\(H_0\)`.<br> Bestimmen des kritischen Werts für einen Test von `\(H_0\)`: `\(\chi^2\)`-Verteilungstest: Verwenden der Exakten Stichprobenverteilung unter `\(H_0\)`: ```r # Anzahl Replikationen Reps <- 9999 # Initialisieren: Vektor der Simulierten Teststatistiken test_stat_sim <- numeric(Reps) ``` --- # 7 Monte Carlo Simulation: ```r for (i in 1:Reps) { # Ziehe zufällig Noten aus Gleichverteilung noten <- c(1,1.3,1.7,2.0,2.3,2.7,3.0,3.3,3.7,4.0,4.3,4.7,5.0) noten.stud <- sample(noten,37,replace=TRUE) # Berechne Häufigkeiten x_sim <- rep(NA,13) for (j in 1:13) x_sim[j] <- sum(noten.stud==noten[j]) # Simulierte Teststatistik test_stat_sim[i] <- sum((x_sim-x0)^2/x0) } ``` Den simulierten P-Value erhält man nun, indem man den Anteil der Simulierten Teststatistiken, die größer sind als die beobachtete Teststatistik, berechnet: ```r mean(test_stat_sim > test_stat) ``` --- # 7 Monte Carlo Ist die `\(\chi^2\)`-Verteilung eine gute Approximation? ```r plot(density(test_stat_sim), main="Simulierte Dichte der Teststatistik unter H0 und Xi^2 Approximation") lines(seq(0,max(test_stat_sim),0.1), dchisq(seq(0,max(test_stat_sim),0.1),df=k), col="orange") ``` --- # 7 Monte Carlo ### Aufgabe 7.1 Verwenden Sie den datengenerierenden Prozess zur Modellselektion von vorher. Führen Sie anstelle des Modellvergleichs mit BIC in jeder Iteration einen statistischen Test auf `\(\beta=0\)` durch und wählen Sie das restringierte Modell, wenn dieser Test abgelehnt wird. Vergleichen Sie das Ergebnis mit dem von vorher. Zeichnen Sie auch diese Verteilung wieder ein. ### Aufgabe 7.2 Führen Sie nun jeweils mit dem gewählten Modell einen Test auf `\(H_0: \alpha=1\)` durch. Führen Sie den gleichen Test im unrestringierten Modell durch. Veranschaulichen Sie das Ergebnis. --- # 8 Numerische Optimierung In VWL, Ökonometrie und Statistik ist häufig Maximierung oder Minimierung von Funktionen gefordert. Bei einigen Funktionen (statistische Zielfunktionen wie Likelihood oder Residuenquadratsumme nichtlinearer Regressionen) ist das Optimierungsproblem analytisch nicht lösbar, es existiert keine (algebraische) Formel für den Schätzer (wie etwa bei OLS). Ausweg: Es ist möglich, die Funktion **numerisch** bzw. allgemeiner **iterativ** (in mehreren Schritten) zu optimieren. Dazu muss i.A. nur der Funktionswert evaluierbar sein. Achtung: Bei numerischen Algorithmen muss auf Eindeutigkeit der Nullstelle, des Optimums, des Fixpunkts usw geachtet werden. U. a. können unterschiedliche Startwerte zu unterschiedlichen Ergebnissen führen (s. unten) --- # 8 Numerische Optimierung In R gibt es verschiedene Funktionen zur numerischen Optimierung - `optimize()`: relativ einfach zu bedienen, kann aber nur univariate Funktionen optimieren - `nlm()`: nichtlineare Optimierung - `optim()`: allgemeines Interface zu mehreren Optimierungsalgorithmen `\(\Rightarrow\)` wir werden hier nur `optim()` behandeln --- # 8 Numerische Optimierung Zur Minimierung benötigt `optim()` folgende Argumente: - `par`: Vektor mit Startwerte von den für die zu minimierende Funktion relevanten Parameter - `fn`: die zu minimierende Funktion optional kann außerdem spezifiziert werden: - `method`: welcher Optimierungsalgorithmus soll verwendet werden? - `"Nelder-Mead"`: etwas langsamer aber dafür relativ robust, auch auf nicht differenzierbare Funktionen anwendbar - `"BFGS"`: benötigt zusätzlich eine erste Ableitung (entweder übergeben mit `gradient` ansonsten numerisch), schneller als `"Nelder-Mead"` - `"CG"`: etwas schneller als `"BFGS"` aber etwas stabiler - `"L-BFGS-B"`: erlaubt Parametereinschränkungen durch obere und untere Grenzen für die Parameter - `"SANN"`: Simulated Annealing, siehe dazu [Eintrag in Wikipedia](https://de.wikipedia.org/wiki/Simulated_Annealing) - `"Brent"`: Für eindimensionale Optimierung, wird auch in `optimize()` verwendet --- # 8 Numerische Optimierung - `lower/upper`: falls `method = "L-BFGS-B"` oder `method = "Brent"`, kann hiermit der Parameterraum eingeschränkt werden - `control`: weitere Kontrollparameter übergeben in einer Liste (z.B. maximale Anzahl der Iterationsschritte) - `hessian`: soll die Hessematrix der Funktion im berechneten Optimum mit ausgegeben werden? Der Rückgabewert der Funktion `optim()` ist eine Liste mit den Einträgen - `par`: Parameterwerte im Optimum - `value`: Funktionswert im Optimum - `counts`: Anzahl der Funktions- und Gradientenaufrufe bis zum Finden des Optimums - `convergence`: 0 falls Optimierung erfolgreich, ansonsten verschiedene Fehlermeldungen --- # 8 Numerische Optimierung ### Einfaches Beispiel ```r f <- function(x) 0.2*(x-1)^4-0.7*x^2-0.3*x+5 curve(f, from = 0, to = 4) ## für eindimensionale Optimierung wähle "Brent" opt <- optim(par=NA, fn=f, lower=-10, upper=10, method="Brent") abline(v = opt$par, col = "orange", lwd = 2, lty = 2) ``` <!-- --> --- # 8 Numerische Optimierung ### Aufgabe 8.1 Modifizieren Sie die Funktion f, so dass vor der Ausgabe des Funktionswerts mit der print-Funktion der x-Wert und der Funktionswert ausgegeben wird. Führen Sie nun die Optimierung durch. Wie erklären Sie sich das? --- # 8 Numerische Optimierung ### Weiteres Beispiel: Nichtlineare Regression (siehe Fortgeschrittene Ök.) ```r ## Regressionsmodell mit zwei Parametern a1 und a2 a <- c( 1 , 0.6 ) n <- 200 x <- rchisq(n,df=3) u <- rnorm(n,sd=0.5) # Nichtlineares Modell y = a x^b+u y <- a[1]*x^a[2] + u # Meist verwendet man stattdessen log(y) = c0 + c1 log(x) + e DATA <- data.frame(y,x) plot(DATA$x,DATA$y) ## Residuenquadratsumme zu minimieren RSS <- function(a,DATA){ u_tilde <- DATA$y - a[1]*DATA$x^a[2] return(sum(u_tilde^2)) } ``` --- # 8 Numerische Optimierung ```r ## Zielfunktion anschauen für verschiedene Werte von a length.grid <- 50 a1_grid <- seq(-1,3,length.out=length.grid) a2_grid <- seq(0,1.5,length.out=length.grid) a_grid <- as.matrix(expand.grid(a1=a1_grid,a2=a2_grid)) RSS_grid <- apply(a_grid, 1, RSS, DATA = DATA) RSS_grid <- matrix(RSS_grid, length.grid, length.grid) # Nicht viel zu sehen contour(a1_grid,a2_grid,RSS_grid,xlab="a1",ylab="a2",nlevels=100) # -> Logarithmieren contour(a1_grid,a2_grid,log(RSS_grid),xlab="a1",ylab="a2",nlevels=100) # 3D-Plot persp(RSS_grid, xlab = "a1", ylab = "a2", phi = 10, theta = -40, ticktype = "detailed") # damit das Minimum später auch Sinn macht: persp(log(RSS_grid), xlab = "a1", ylab = "a2", phi = 10, theta = -40, ticktype="detailed") ``` --- # 8 Numerische Optimierung ### Numerische Minimierung ```r est <- optim(par = c(1,1), fn = RSS, DATA = DATA) points(x = est$par[1], y = est$par[2] , col = "red" , pch = 20) ``` Fazit: <br> Am Tripel `\((0.99, 0.61, 45.14) = (a_1, a_2, f(a_1,a_2))\)` liegt das Minimum der Residuenquadratsumme, da am Punkt `\((a_1, a_2)\)` die Funktion RSS das Minimum, den geschätzten Wert 45.14 annimmt<br> Allgemein:<br> Hat man eine Funktion `\(f: \mathbb{R}^n \rightarrow \mathbb{R}^n\)`, so hat der Graph Dimension `\(n+m\)`. In diesem Fall: `\(\mathbb{R}^m=\mathbb{R}^2\)` und `\(\mathbb{R}^n=\mathbb{R}\)`, also ist der Graph dreidimensional. Beim vorigen Beispiel ging es um die Berechnung eines nichtlinearen Kleinst-Quadrate-Schätzers, hierfür gibt es in R eine eigene Funktion: ```r est_nls <- nls(y ~ a1 * x^a2, start = c(a1 = 1,a2 = 1), data = DATA) summary(est_nls) ``` --- # 8 Numerische Optimierung ### Startwerten haben einen Einfluss auf das Optimum: ```r # Ziel: Maximieren der Funktion f f <- function(x) (2*cos(x[1]/0.5)-0.3*x[1]^2)+cos(x[2]^2)-0.5*(x[2]-2)^2 # max statt min: control-Argument benutzen (oder g=-f minimieren) optim(par=c(1,1),f,control=list(fnscale=-1)) optim(par=c(-0.5,3),f,control=list(fnscale=-1)) # Andere Startwerte... ``` --- # 8 Numerische Optimierung ```r # Funktion betrachten l.grid <- 50 grid.x <- as.matrix(expand.grid(x1 = seq(-5,5,length.out=l.grid), x2 = seq(-5,5,length.out=l.grid))) grid.f <-apply(grid.x, 1, f) matrix.grid.f <- matrix(grid.f ,l.grid) image(matrix.grid.f,xlab="x1",ylab="x2") contour(matrix.grid.f,nlevels=100,add=TRUE) persp(x = seq(-5,5,length.out=l.grid), y = seq(-5,5,length.out=l.grid), z = matrix.grid.f, xlab = "x1", ylab = "x2", theta = 60, ticktype = "detailed") # Und Startwerte geschickt wählen (x.start <- grid.x[which.max(grid.f),]) optim(par=x.start,f,control=list(fnscale=-1)) ``` --- # 8 Numerische Optimierung ### Aufgabe 8.2 Maximieren Sie die Funktion f ```r f <- function(x) (2 * cos( x[1]/0.5 ) - 0.3*x[1]^2) + cos(x[2]^2) - 0.5 * (x[2] - 2)^2 ``` Gehen Sie davon aus, dass das Maximum (x1, x2) -5<x1<5 und -5<x2<5 erfüllt. Achten Sie auf die Startwerte! --- # 8. Numerische Optimierung ### Weiteres Beispiel: Maximum Likelihood Schätzung GARCH-Modell `$$x_t = h_t \cdot \varepsilon_t$$` `$$\varepsilon_t \sim IID(0,1) \text{ (White Noise)}$$` `$$\operatorname{Var}(x_t | \mathcal{I}_{t-1}) = h_t^2$$` Bedingte Varianz ist dabei von der Vorperiode abhängig: `$$h_t^2 = \alpha + \gamma_0 h_{t-1}^2 + \dots + \gamma_q h_{t-q}^2 + \beta_1 \varepsilon_{t-1}^2 + \dots + \beta_p \varepsilon_{t-p}^2$$` Wir brauchen nun eine Likelihood-Funktion, die von den Parametern `theta` (Vektor, der alle Parameter des Modells zusammenfasst) und von den Daten `x` (univariate Zeitreihe) abhängt. --- # 8. Numerische Optimierung ```r LogLik.GARCH <- function(theta, x){ ## GARCH(1,1) c <- theta[1] # Konstante in GARCH Gleichung a <- theta[2] # ARCH Parameter g <- theta[3] # GARCH Parameter # Unzulässige Parameterwerte ausschließen (-> Inf als Wert) if (sum(a) + sum(g) > 1) return(NA) # Instabil -> nicht zugelassen if (any(theta < 0)) return(NA) # Nichtnegativität verletzt -> nicht zugelassen n <- length(x) # Stichprobengröße h_sq <- rep(var(x),n) # Vektor der bedingten Varianzen (am Anfang: unbedingte Var.) ll <- numeric(n) # Vektor der (negativen) Log Likelihoods pro Beobachtung # Iterativ Bedingte Varianz und negative Likelihood berechnen for (i in 2:n){ h_sq[i] <- c + a * x[i-1]^2 + g * h_sq[i-1] ll[i] <- - 0.5 * (log(2*pi) + log(h_sq[i]) + x[i]^2/h_sq[i]) # negative Likelihood } # Funktionswert = (negative) Log Likelihood return(sum(ll)) } ``` --- # 8. Numerische Optimierung Speichere hierzu zunächst Datei [wheat.txt](https://www.uni-regensburg.de/wirtschaftswissenschaften/vwl-tschernig/medien/programmieren-mit-r/wheat.txt) lokal unter `wheat.txt` ab. ```r wheat <- read.table("wheat.txt",header=TRUE) %>% na.omit() ## Startwerte festlegen par_start <- c(var(wheat$Change)*0.05 , 0.5 , 0.5) ## Test Funktionsaufruf: LogLik.GARCH( par_start , x = wheat$Change) ## Optimum berechnen opt <- optim( par_start , LogLik.GARCH , x = wheat$Change , control = list(fnscale = -1 , maxit = 10000) ) ``` --- # 8. Numerische Optimierung ### Aufgabe 8.3 Im der Datei [FunktMIN.RData](https://www.uni-regensburg.de/wirtschaftswissenschaften/vwl-tschernig/medien/programmieren-mit-r/funktmin.rdata) sind die Vektoren `y`, `x` und `z` gespeichert. Betrachten Sie das Regressionsmodell `$$y = \beta_1+\beta_2\, x + \beta_3\, z^{\beta_4} + u$$` 1. Geben Sie eine Funktion an, die aus den Daten und beliebigen Werten von `beta` die Residuenquadratsumme berechnet. 2. Berechnen Sie den Wert von beta, der diese SSR minimiert. 3. Versuchen Sie, die Funktion `f` in der Datei `FunktMIN.RData` zu minimieren. Geben Sie auch die Hessematrix an. --- # 9 Bootstrap ### Grundlagen: Das Bootstrap-Verfahren ist eine *Resampling-Methode* mit dem Ziel, unbekannte Test-Verteilungen zu schätzen. Die Idee ist dabei, aus den beobachteten Daten durch Ziehen mit Zurücklegen neue (künstliche) Stichproben zu generieren und aus jeder Stichprobe eine Teststatistik berechnen. Mit diesen Teststatistiken lässt sich dann die Verteilung der Teststatistik schätzen und damit P-Werte des Tests berechnen. Siehe dazu z.B. [Skript zum Kurs Methoden der Ökonometrie](https://www.uni-regensburg.de/wirtschaftswissenschaften/vwl-tschernig/medien/methoden-der-oekonometrie/methoden_oekonometrie_handout_2018_11_29.pdf), Kapitel 11.5. --- # 9 Bootstrap ### Beispiel (Parametertest AR(1)-Modell) Simuliere `\(AR(1)\)`-Prozess mit nicht normalverteilten Fehlern: `$$y_t = \alpha y_{t-1} + u_t,\ u_t\sim t(5)$$` Simuliere Daten: ```r ## Parameter set.seed(400) a <- 0.9 n <- 150 u <- rt(n,df=5) ## Berechnet Zeitreihe aus Fehlern y <- stats::filter(u,a,"recursive") ## filter explizit aus stats! ``` --- # 9 Bootstrap Schätzung des AR(1)-Parameters und *asymptotischer* Test auf `\(\alpha = 0.9\)`: ```r library(dynlm) est.ar <- dynlm(y~L(y,1)) ## Asymptotischer Test t_stat <- (summary(est.ar)$coef[2,1]-0.9)/summary(est.ar)$coef[2,2] pnorm(abs(t_stat),lower.tail=FALSE)*2 ``` Die genutzte Testverteilung ist nur asymptotisch korrekt und für endliche Stichproben lediglich näherungsweise gültig. Unter bestimmten Voraussetzungen ist eine Bootstrap-Approximation besser. Vorgehen:<br> Ziehe aus Residuen mit Zurücklegen und generiere Zeitreihen unter der `\(H_0\)`, also `\(\alpha=0.9\)`. Berechne dann jeweils die Teststatistik. --- # 9 Bootstrap ```r (u_hat <- est.ar$resid) Reps <- 999 a_sim <- numeric(Reps) t_sim <- numeric(Reps) for (i in 1:Reps){ u_sim <- sample(u_hat, n, replace=TRUE) # Ziehen aus den Residuen (mit Zurücklegen!) y_sim <- stats::filter(u_sim, 0.9, "recursive") # Nullhypothesenwert verwenden! est_sim <- dynlm(y_sim ~ L(y_sim,1)) a_sim[i] <- est_sim$coef[2] t_sim[i] <- (a_sim[i]-0.9)/summary(est_sim)$coef[2,2] } # Berechne P-Value mean(abs(t_sim)>abs(t_stat)) plot(density(t_sim), main="Asymptotic and bootstrap distribution of t", xlab="t-Statistic") lines(seq(-4,4,0.1), dnorm(seq(-4,4,0.1)), col="orange") legend("topright", c("Bootstrap", "Asymptotic"), lty=c(1,1), col=c(1,"orange")) ``` --- # 9 Bootstrap <!-- --> --- # 9 Bootstrap ### Aufgaben 9.1 Wiederholen Sie die Bootstrap-Prozedur im AR(1) (oder einem anderen) Beispiel Führen Sie jeweils einen Bootstrap- und einen asymptotischen (t-) Test bzgl. `\(\alpha\)` durch. Wie gut ist der Bootstrap bei wahrer Nullhypothese? ### Aufgaben 9.2 Plotten Sie die Ablehnungshäufigkeiten des Bootstrap- und asymptotischen Tests in Abhängigkeit von `\(\alpha\)`. --- # 10 Effizienz In diesem Kapitel werden einige Hinweise zur Programmierung allgemein und zur Laufzeiteffizienz Allgemein:<br> - überlege einheitliche Ordnerstruktur für Daten, R-Skripte, Graphiken, Outputs,... - ideal: R-Paket erstellen, da hier schon Dateistruktur vorgegeben - Es gibt Versionskontrollsysteme, damit lassen sich verschiedene Zustände von Codeversionen festhalten und es ist im Nachhinein immer klar, was wann verändert wurde. Beispiel [git](https://git-scm.com/). - Kommentieren von Code extrem wichtig, sonst häufig später nicht mehr lesbar! --- # 10 Effizienz ### Aufgabe 10.1 Versuchen Sie, den nachfolgenden Code zu verstehen. Bringen Sie ihn dabei in eine Form, in der dies einfacher ist. (Code entzerren, Strukturieren, Variablennamen ändern, Kommentieren) ```r f <- function(a05,a1,a2,a3,a4,a100,a1000) { fi <- function(b05,b1,b2, b3,b4,b100,b123){d <- list();d$x <- rnorm(b100); set.seed(b123);d$y=d$x*b1+rt(b100,df=b4)*exp(b2+b3*d$x); d=as.data.frame(d); library(AER);h <- paste("d$x=",b1, sep="");(linearHypothesis(lm(d$y~d$x),h)[[6]][2]<b05)} a <- numeric();for (i in 1:a1000) a[i] <- fi(a05,a1,a2,a3 ,a4,a100,i);mean(a)} f(0.05,1,1,1,4,100,1000) ``` --- # 10 Effizienz ### Einige Regeln für effizienten Code - Vektorisieren! Dann werden nämlich Schleifen in C genutzt und nicht Schleifen in R - Objekte nicht nach und nach größer machen (z.B. in Schleife), immer erst Leerobjekt initialisieren und dann befüllen - Einige R-Funktionen haben, um mit allen möglichen Fällen umgehen zu können, einen relativ großen *overhead*. Meistens gibt es weniger allgemeine, dafür schnellere Funktionen, die dann auch allerdings weniger mächtig sind. Zum Beispiel: - `lm()` vs. `lm.fit()` oder `.lm.fit()` - Um herauszufinden, an welcher Stelle viel Laufzeit benötigt wird und wo es sich ggf. lohnt zu optimieren, existierten Profiling-Methoden - Man muss aber abwägen: schnellere Laufzeit vs. zusätzlichh benötigte Zeit zum Implementieren --- # 10 Effizienz --- # Thanks! .center[ Slides created via the R package [**xaringan**](https://github.com/yihui/xaringan). The chakra comes from [remark.js](https://remarkjs.com), [**knitr**](http://yihui.name/knitr), and [R Markdown](https://rmarkdown.rstudio.com). ]